Кривая роста бактерий (логистическая / сигмовидная) с несколькими объясняющими переменными в R
Цель: я хочу получить регрессию (кривые ggplot и параметры модели) для кривых роста с несколькими обработками.
У меня есть данные о бактериальных культурах C={a,b,c,d}, растущих на источниках питательных веществ N={x,y}.
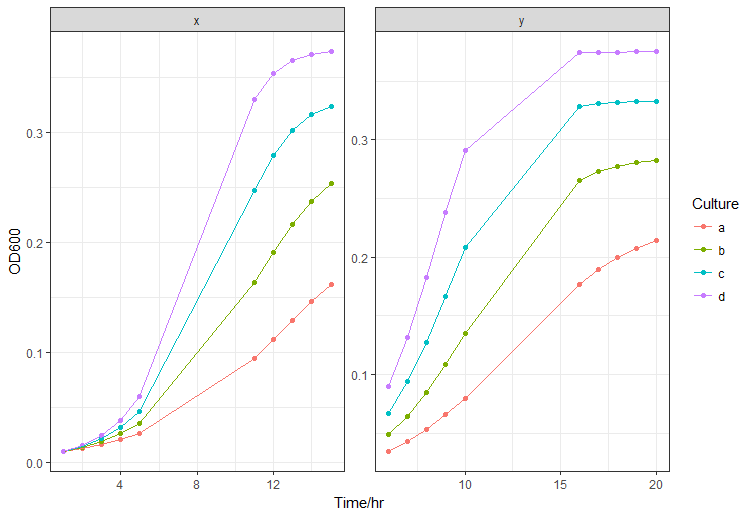
Их идеализированные кривые роста (измерения мутности клеточной культуры каждый час) выглядят примерно так: 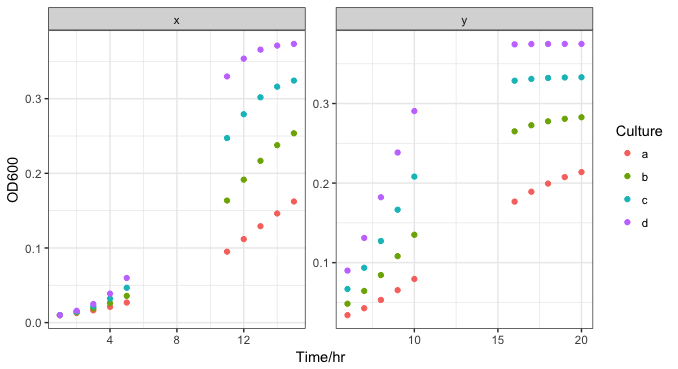
Есть 8 различных кривых для получения коэффициентов и кривых для. Как я могу сделать это за один раз для моего фрейма данных, используя разные методы лечения для нелинейной регрессии?
Спасибо!!!
Этот вопрос похож на оставшийся без ответа вопрос, размещенный здесь.
(исходный код для идеализированных данных, извините, это не элегантно, так как я не ученый):
a<-1:20
a[1]<-0.01
for(i in c(1:19)){
a[i+1]<-1.3*a[i]*(1-a[i])
}
b<-1:20
b[1]<-0.01
for(i in c(1:19)){
b[i+1]<-1.4*b[i]*(1-b[i])
}
c<-1:20
c[1]<-0.01
for(i in c(1:19)){
c[i+1]<-1.5*c[i]*(1-c[i])
}
d<-1:20
d[1]<-0.01
for(i in c(1:19)){
d[i+1]<-1.6*d[i]*(1-d[i])
}
sub.data<-cbind(a,b,c,d)
require(reshape2)
data<-melt(sub.data, value.name = "OD600")
data$nutrition<-rep(c("x", "y"), each=5, times=4)
colnames(data)[1:2]<-c("Time", "Culture")
ggplot(data, aes(x = Time, y = OD600, color = Culture, group=nutrition)) +
theme_bw() + xlab("Time/hr") + ylab("OD600") +
geom_point() + facet_wrap(~nutrition, scales = "free")
1 ответ
Если вы знакомы group_by функция от dplyr (включен в tidyverse), то вы можете сгруппировать данные по культуре и питанию и создать модели для каждой группы, используя broom, Я думаю, что эта виньетка дает именно то, что вы пытаетесь достичь. Вот код все сразу:
library(tidyverse)
library(broom)
library(mgcv) #For the gam model
data %>%
group_by(Culture, nutrition) %>%
do(fit = gam(OD600 ~ s(Time), data = ., family=gaussian())) %>% # Change this to whatever model you want (e.g., non-linear regession, sigmoid)
#do(fit = lm(OD600 ~ Time, data = .,)) %>% # Example using linear regression
augment(fit) %>%
ggplot(aes(x = Time, y = OD600, color = Culture)) + # No need to group by nutrition because that is broken out in the facet_wrap
theme_bw() + xlab("Time/hr") + ylab("OD600") +
geom_point() + facet_wrap(~nutrition, scales = "free") +
geom_line(aes(y = .fitted, group = Culture))
Если ты в порядке без одного шага, разбей на части %>% для лучшего понимания. Я использовал GAM, который здесь подходит, но вы можете заменить его на любую модель, какую захотите, включая сигмовидную.