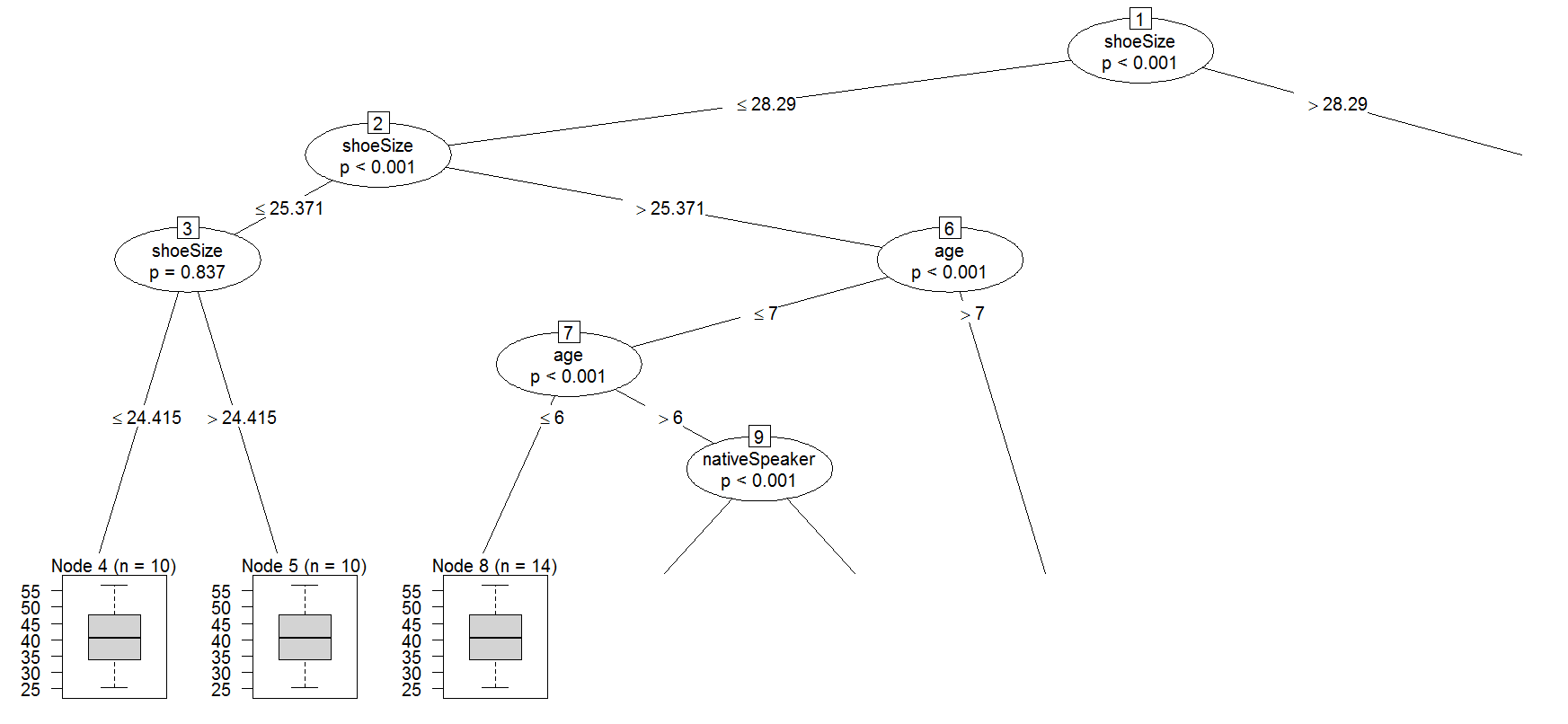
cforest печатает пустое дерево
Я пытаюсь использовать функцию cforest (R, party package).
Вот что я делаю, чтобы построить лес:
library("party")
set.seed(42)
readingSkills.cf <- cforest(score ~ ., data = readingSkills,
control = cforest_unbiased(mtry = 2, ntree = 50))
Затем я хочу напечатать первое дерево, и я делаю
party:::prettytree(readingSkills.cf@ensemble[[1]],names(readingSkills.cf@data@get("input")))
Результат выглядит так
1) shoeSize <= 28.29018; criterion = 1, statistic = 89.711
2) age <= 6; criterion = 1, statistic = 48.324
3) age <= 5; criterion = 0.997, statistic = 8.917
4)* weights = 0
3) age > 5
5)* weights = 0
2) age > 6
6) age <= 7; criterion = 1, statistic = 13.387
7) shoeSize <= 26.66743; criterion = 0.214, statistic = 0.073
8)* weights = 0
7) shoeSize > 26.66743
9)* weights = 0
6) age > 7
10)* weights = 0
1) shoeSize > 28.29018
11) age <= 9; criterion = 1, statistic = 36.836
12) nativeSpeaker == {}; criterion = 0.998, statistic = 9.347
13)* weights = 0
12) nativeSpeaker == {}
14)* weights = 0
11) age > 9
15) nativeSpeaker == {}; criterion = 1, statistic = 19.124
16) age <= 10; criterion = 1, statistic = 18.441
17)* weights = 0
16) age > 10
18)* weights = 0
15) nativeSpeaker == {}
19)* weights = 0
Почему он пуст (вес в каждом узле равен нулю)?
2 ответа
Краткий ответ: вес корпуса weights в каждом узле есть NULLне хранится prettytree функциональные выходы weights = 0, поскольку sum(NULL) равно 0 в R.
Рассмотрим следующее ctree пример:
library("party")
x <- ctree(Species ~ ., data=iris)
plot(x, type="simple")
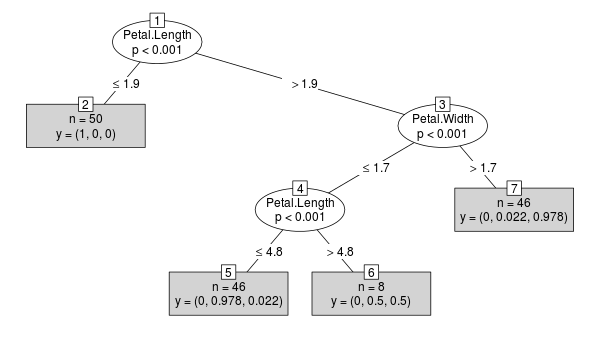
Для полученного объекта x (учебный класс BinaryTree) весовые коэффициенты хранятся в каждом узле:
R> sum(x@tree$left$weights)
[1] 50
R> sum(x@tree$right$weights)
[1] 100
R> sum(x@tree$right$left$weights)
[1] 54
R> sum(x@tree$right$right$weights)
[1] 46
Теперь давайте внимательнее посмотрим на cforest:
y <- cforest(Species ~ ., data=iris, control=cforest_control(mtry=2))
tr <- party:::prettytree(y@ensemble[[1]], names(y@data@get("input")))
plot(new("BinaryTree", tree=tr, data=y@data, responses=y@responses))
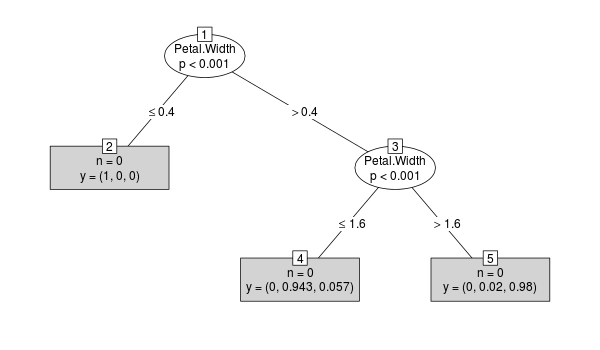
Веса кейсов не сохраняются в ансамбле дерева, что можно увидеть по следующему:
fixInNamespace("print.TerminalNode", "party")
изменить print метод для
function (x, n = 1, ...)·
{
print(names(x))
print(x$weights)
cat(paste(paste(rep(" ", n - 1), collapse = ""), x$nodeID,·
")* ", sep = "", collapse = ""), "weights =", sum(x$weights),·
"\n")
}
Теперь мы можем наблюдать, что weights является NULL в каждом узле:
R> tr
1) Petal.Width <= 0.4; criterion = 10.641, statistic = 10.641
[1] "nodeID" "weights" "criterion" "terminal" "psplit"
[6] "ssplits" "prediction" "left" "right" NA
NULL
2)* weights = 0
1) Petal.Width > 0.4
3) Petal.Width <= 1.6; criterion = 8.629, statistic = 8.629
[1] "nodeID" "weights" "criterion" "terminal" "psplit"
[6] "ssplits" "prediction" "left" "right" NA
NULL
4)* weights = 0
3) Petal.Width > 1.6
[1] "nodeID" "weights" "criterion" "terminal" "psplit"
[6] "ssplits" "prediction" "left" "right" NA
NULL
5)* weights = 0
Обновите это хак, чтобы отобразить суммы весов кейсов:
update_tree <- function(x) {
if(!x$terminal) {
x$left <- update_tree(x$left)
x$right <- update_tree(x$right)
} else {
x$weights <- x[[9]]
x$weights_ <- x[[9]]
}
x
}
tr_weights <- update_tree(tr)
plot(new("BinaryTree", tree=tr_weights, data=y@data, responses=y@responses))
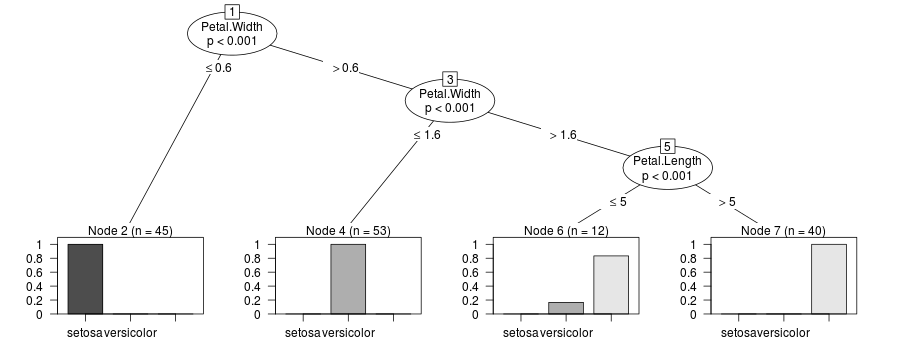
Решение, предложенное @rcs в обновлении, интересно, но не работает с cforest когда зависимая переменная является числовой. Код:
set.seed(12345)
y <- cforest(score ~ ., data = readingSkills,
control = cforest_unbiased(mtry = 2, ntree = 50))
tr <- party:::prettytree(y@ensemble[[1]], names(y@data@get("input")))
tr_weights <- update_tree(tr)
plot(new("BinaryTree", tree=tr_weights, data=y@data, responses=y@responses))
генерирует следующее сообщение об ошибке
R> Error in valid.data(rep(units, length.out = length(x)), data) :
no string supplied for 'strwidth/height' unit
и следующий сюжет:
Ниже я предлагаю улучшенную версию взлома, предложенного @rcs:
get_cTree <- function(cf, k=1) {
dt <- cf@data@get("input")
tr <- party:::prettytree(cf@ensemble[[k]], names(dt))
tr_updated <- update_tree(tr, dt)
new("BinaryTree", tree=tr_updated, data=cf@data, responses=cf@responses,
cond_distr_response=cf@cond_distr_response, predict_response=cf@predict_response)
}
update_tree <- function(x, dt) {
x <- update_weights(x, dt)
if(!x$terminal) {
x$left <- update_tree(x$left, dt)
x$right <- update_tree(x$right, dt)
}
x
}
update_weights <- function(x, dt) {
splt <- x$psplit
spltClass <- attr(splt,"class")
spltVarName <- splt$variableName
spltVar <- dt[,spltVarName]
spltVarLev <- levels(spltVar)
if (!is.null(spltClass)) {
if (spltClass=="nominalSplit") {
attr(x$psplit$splitpoint,"levels") <- spltVarLev
filt <- spltVar %in% spltVarLev[as.logical(x$psplit$splitpoint)]
} else {
filt <- (spltVar <= splt$splitpoint)
}
x$left$weights <- as.numeric(filt)
x$right$weights <- as.numeric(!filt)
}
x
}
plot(get_cTree(y, 1))