Как лучше переназначить коллекцию файлов NetCDF в набор данных Zarr
Я пытаюсь переназначить коллекцию файлов NetCDF и создать набор данных Zarr на AWS S3. У меня есть 168 оригинальных классических файлов NetCDF4 с массивами измерений time: 1, y: 3840, x: 4608 порванный как chunks={'time':1, 'y':768, 'x':922},
Я хочу записать этот вывод в Zarr, и я хочу оптимизировать его для извлечения временных рядов, поэтому включайте больше временных записей в мои куски. Я подумал, что буду использовать xarray, чтобы помочь выполнить работу, так как у меня есть много процессоров, доступных для использования Dask, и xarray имеет оба xr.open_mfdataset а также ds.to_zarr,
Я сначала попытался повторить chunks={'time':24, 'y':768, 'x':922} чтобы соответствовать входной NetCDF4 порции в x а также y, но когда я попытался написать Zarr, он пожаловался, потому что требовался одинаковый размер куска в обоих x а также yдопускается только неравномерный размер в последнем фрагменте вдоль time измерение (и, к сожалению, в x размер, общий размер 4608 не кратен размеру куска 922.
Итак, я попытался chunks={'time':168, 'y':384, 'x':288} и это начало работать, и продолжалось очень быстро в течение нескольких минут, затем становилось все медленнее и медленнее. Через 50 минут кластер умер с:
4072 distributed.core - INFO - Event loop was unresponsive in Worker for 1.41s. This is often caused by long-running GIL-holding functions or moving large chunks of data. This can cause timeouts and instability.
4073 slurmstepd: error: Step 3294889.0 exceeded memory limit (25346188 > 25165824), being killed
Вот код, который я использую:
from dask.distributed import Client
import pandas as pd
import xarray as xr
import s3fs
import zarr
client = Client(scheduler_file='/home/rsignell/scheduler.json')
client
root = '/lustre/projects/hazards/cmgp/woodshole/rsignell/nwm/forcing_short_range/'
bucket_endpoint='https://s3.us-west-1.amazonaws.com/'
f_zarr = 'rsignell/nwm/test_week4'
dates = pd.date_range(start='2018-04-01T00:00', end='2018-04-07T23:00', freq='H')
urls = ['{}{}/nwm.t{}z.short_range.forcing.f001.conus.nc'.format(root,a.strftime('%Y%m%d'),a.strftime('%H')) for a in dates]
ds = xr.open_mfdataset(urls, concat_dim='time', chunks={'time':1, 'y':768, 'x':922})
ds = ds.drop(['ProjectionCoordinateSystem','time_bounds'])
ds = ds.chunk(chunks={'time':168, 'y':384, 'x':288}).persist()
ds
производства
<xarray.Dataset>
Dimensions: (reference_time: 168, time: 168, x: 4608, y: 3840)
Coordinates:
* reference_time (reference_time) datetime64[ns] 2018-04-01 ...
* x (x) float64 -2.304e+06 -2.303e+06 -2.302e+06 -2.301e+06 ...
* y (y) float64 -1.92e+06 -1.919e+06 -1.918e+06 -1.917e+06 ...
* time (time) datetime64[ns] 2018-04-01T01:00:00 ...
Data variables:
T2D (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
LWDOWN (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
Q2D (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
U2D (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
V2D (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
PSFC (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
RAINRATE (time, y, x) float32 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
SWDOWN (time, y, x) float64 dask.array<shape=(168, 3840, 4608), chunksize=(168, 384, 288)>
Тогда я звоню
fs = s3fs.S3FileSystem(anon=False, client_kwargs=dict(endpoint_url=bucket_endpoint))
d = s3fs.S3Map(f_zarr, s3=fs)
compressor = zarr.Blosc(cname='zstd', clevel=3, shuffle=2)
encoding = {vname: {'compressor': compressor} for vname in ds.data_vars}
delayed_store = ds.to_zarr(store=d, mode='w', encoding=encoding, compute=False)
persist_store = delayed_store.persist(retries=100)
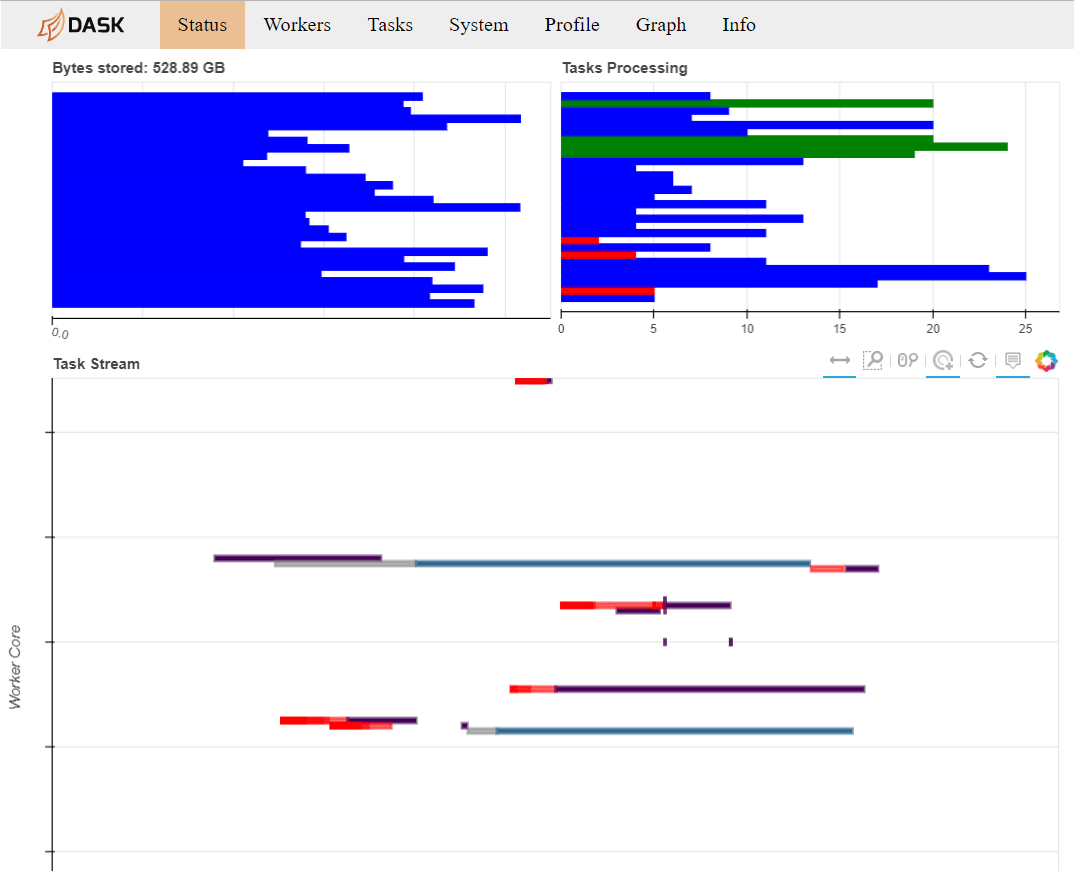
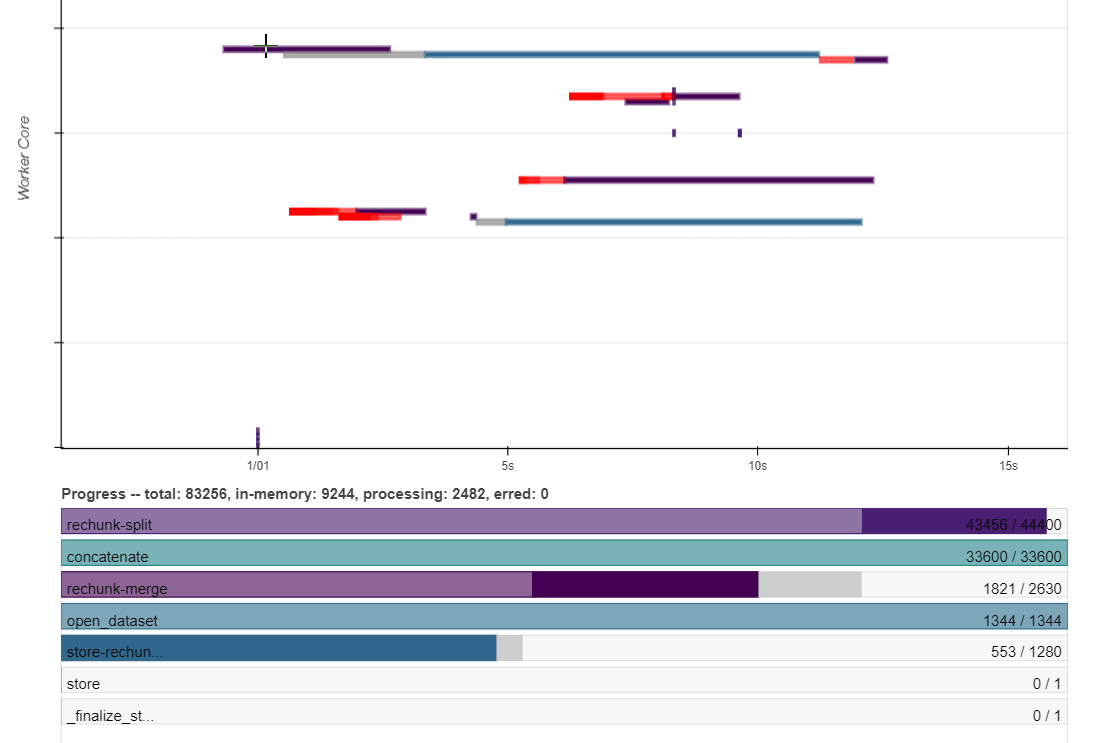
и как раз перед смертью Dask Daskboard выглядит так:
Общий размер файлов NetCDF4 составляет 20 ГБ, поэтому кажется немного сумасшедшим, что у меня есть более 500 ГБ, отображаемых в Dask Dashboard, и что 30 процессоров каждый с 60 ГБ оперативной памяти не достаточно для этой работы.
Что я делаю не так, или что будет лучшим решением?
0 ответов
Я заметил, что вы говорите, что хотите увеличить количество фрагментов во временном измерении. А может я неправильно понял.
Вы начинаете с кусков, указанных как chunks={'time':1, 'y':768, 'x':922}, но тогда попробуйте chunks={'time':168, 'y':384, 'x':288} и обнаруживаем, что второй использует большой объем памяти.
Проблема в том, что chunksключевое слово определяет размер фрагментов, а не количество фрагментов!
В первом случае размер каждого фрагмента равен 1*768*922 ~ 7e5, тогда как во втором случае размер каждого фрагмента равен 168*384*288 ~ 2e7.
Максимальное количество порций по времени достигается за счет chunks={'time': 1}.