Одинокая загрузка csv ТОО медленная по сравнению с Matlab
Я разместил этот вопрос, потому что мне было интересно, сделал ли я что-то ужасно неправильное, чтобы получить этот результат.
У меня есть CSV-файл среднего размера, и я попытался использовать NumPy, чтобы загрузить его. Для иллюстрации я сделал файл, используя python:
import timeit
import numpy as np
my_data = np.random.rand(1500000, 3)*10
np.savetxt('./test.csv', my_data, delimiter=',', fmt='%.2f')
И затем я попробовал два метода: numpy.genfromtxt, numpy.loadtxt
setup_stmt = 'import numpy as np'
stmt1 = """\
my_data = np.genfromtxt('./test.csv', delimiter=',')
"""
stmt2 = """\
my_data = np.loadtxt('./test.csv', delimiter=',')
"""
t1 = timeit.timeit(stmt=stmt1, setup=setup_stmt, number=3)
t2 = timeit.timeit(stmt=stmt2, setup=setup_stmt, number=3)
И результат показывает, что t1 = 32,159652940464184, t2 = 52.00093725634724.
Тем не менее, когда я попытался использовать Matlab:
tic
for i = 1:3
my_data = dlmread('./test.csv');
end
toc
Результат показывает: истекшее время составляет 3,196465 секунд.
Я понимаю, что могут быть некоторые различия в скорости загрузки, но:
- Это намного больше, чем я ожидал;
- Разве не np.loadtxt должен быть быстрее, чем np.genfromtxt?
- Я еще не пробовал Python CSV-модуль, потому что загрузка CSV-файла - это очень частая вещь, которую я делаю, а с модулем CSV кодирование немного многословно... Но я был бы рад попробовать, если это единственный способ, В настоящее время меня больше беспокоит то, что я делаю что-то не так.
Любой вклад будет оценен. Заранее большое спасибо!
4 ответа
Да, читаю csv файлы в numpy довольно медленно В пути кода много чистого Python. В эти дни, даже когда я использую чистый numpy Я все еще использую pandas для IO:
>>> import numpy as np, pandas as pd
>>> %time d = np.genfromtxt("./test.csv", delimiter=",")
CPU times: user 14.5 s, sys: 396 ms, total: 14.9 s
Wall time: 14.9 s
>>> %time d = np.loadtxt("./test.csv", delimiter=",")
CPU times: user 25.7 s, sys: 28 ms, total: 25.8 s
Wall time: 25.8 s
>>> %time d = pd.read_csv("./test.csv", delimiter=",").values
CPU times: user 740 ms, sys: 36 ms, total: 776 ms
Wall time: 780 ms
В качестве альтернативы, в достаточно простом случае, подобном этому, вы можете использовать что-то вроде того, что написал здесь Джо Кингтон:
>>> %time data = iter_loadtxt("test.csv")
CPU times: user 2.84 s, sys: 24 ms, total: 2.86 s
Wall time: 2.86 s
Есть также библиотека для чтения текста Уоррена Векессера, на случай, если pandas слишком тяжелая зависимость:
>>> import textreader
>>> %time d = textreader.readrows("test.csv", float, ",")
readrows: numrows = 1500000
CPU times: user 1.3 s, sys: 40 ms, total: 1.34 s
Wall time: 1.34 s
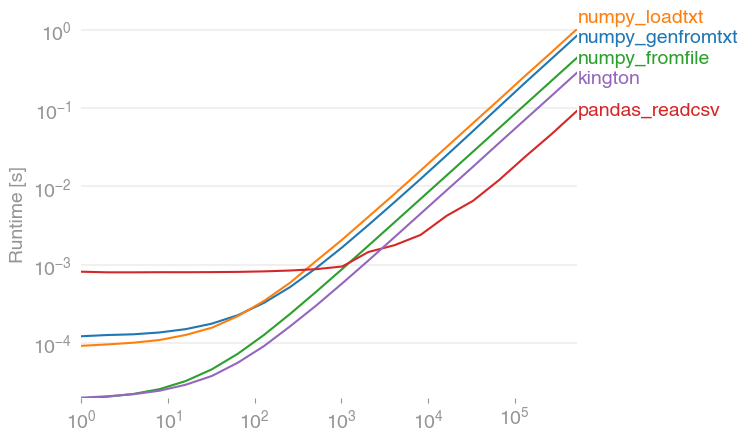
Я протестировал предлагаемые решения с помощью perfplot ( мой небольшой проект) и обнаружил, что
pandas.read_csv(filename)
действительно самое быстрое решение (если прочитано более 2000 записей, до этого все находится в диапазоне миллисекунд). Он превосходит варианты numpy примерно в 10 раз (numpy.fromfile здесь только для сравнения, он не может читать реальные файлы csv.)
Код для воспроизведения сюжета:
import numpy
import pandas
import perfplot
numpy.random.seed(0)
filename = "a.txt"
def setup(n):
a = numpy.random.rand(n)
numpy.savetxt(filename, a)
return None
def numpy_genfromtxt(data):
return numpy.genfromtxt(filename)
def numpy_loadtxt(data):
return numpy.loadtxt(filename)
def numpy_fromfile(data):
out = numpy.fromfile(filename, sep=" ")
return out
def pandas_readcsv(data):
return pandas.read_csv(filename, header=None).values.flatten()
def kington(data):
delimiter = " "
skiprows = 0
dtype = float
def iter_func():
with open(filename, 'r') as infile:
for _ in range(skiprows):
next(infile)
for line in infile:
line = line.rstrip().split(delimiter)
for item in line:
yield dtype(item)
kington.rowlength = len(line)
data = numpy.fromiter(iter_func(), dtype=dtype).flatten()
return data
perfplot.show(
setup=setup,
kernels=[numpy_genfromtxt, numpy_loadtxt, numpy_fromfile, pandas_readcsv, kington],
n_range=[2 ** k for k in range(20)],
logx=True,
logy=True,
)
Если вы хотите просто сохранить и прочитать пустой массив, гораздо лучше сохранить его как двоичный или сжатый двоичный файл в зависимости от размера:
my_data = np.random.rand(1500000, 3)*10
np.savetxt('./test.csv', my_data, delimiter=',', fmt='%.2f')
np.save('./testy', my_data)
np.savez('./testz', my_data)
del my_data
setup_stmt = 'import numpy as np'
stmt1 = """\
my_data = np.genfromtxt('./test.csv', delimiter=',')
"""
stmt2 = """\
my_data = np.load('./testy.npy')
"""
stmt3 = """\
my_data = np.load('./testz.npz')['arr_0']
"""
t1 = timeit.timeit(stmt=stmt1, setup=setup_stmt, number=3)
t2 = timeit.timeit(stmt=stmt2, setup=setup_stmt, number=3)
t3 = timeit.timeit(stmt=stmt3, setup=setup_stmt, number=3)
genfromtxt 39.717250824
save 0.0667860507965
savez 0.268463134766
Возможно, лучше установить простой код на c, который преобразует данные в двоичный файл и позволяет "numpy" читать двоичный файл. У меня есть CSV-файл 20 ГБ для чтения с данными CSV, представляющими собой смесь int, double, str. Простое чтение структур в массив занимает больше часа, в то время как выгрузка в двоичный файл занимает около 2 минут, а загрузка в numpy занимает менее 2 секунд!
Мой конкретный код, например, доступен здесь.
FWIW встроенный модуль CSV прекрасно работает и на самом деле не так многословно.
CSV-модуль:
%%timeit
with open('test.csv', 'r') as f:
np.array([l for l in csv.reader(f)])
1 loop, best of 3: 1.62 s per loop
np.loadtext:
%timeit np.loadtxt('test.csv', delimiter=',')
1 loop, best of 3: 16.6 s per loop
pd.read_csv:
%timeit pd.read_csv('test.csv', header=None).values
1 loop, best of 3: 663 ms per loop
Лично мне нравится использовать панд read_csv но модуль CSV хорош, когда я использую чистый NumPy.