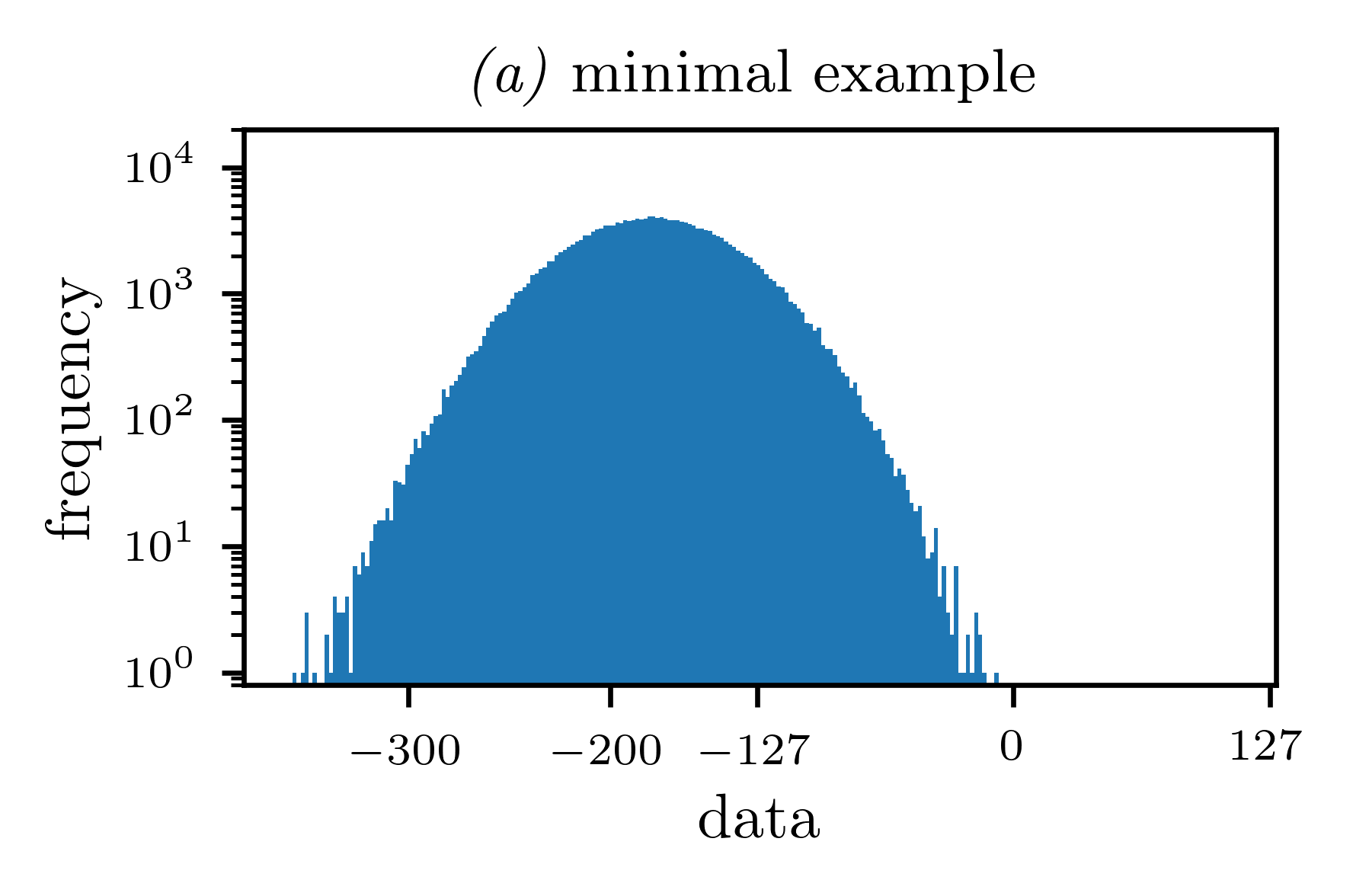
Matplotlib 2.0 полосы в гистограмме
Я получаю вертикальные полосы между бинами при создании гистограммы с помощью matplotlib 2.0.2, python2.7, Win7,64bit, видимых как в созданных pdf, так и в png. Я использую pgf с латексом для создания PDF, который я буду использовать с помощью includegraphics в документе pdflatex. Созданный PNG - это просто быстрая проверка.
Это было не так в Matplotlib 1.5.3. Как мне избавиться от этих белых линий, разделяющих отдельные корзины?
Вещи пытались:
- Включение / выключение сглаживания (aa=True/False в исторической команде)
- рисование линии (ls="-"/ls="none" в исторической команде)
- Одной из вещей, которая могла бы работать, является задание ширины ячеек (ширина =2,3), но это также не работает для PDF при всех значениях масштаба.
Код для создания изображения
import matplotlib as mpl
mpl.use('pgf')
pgf_with_latex = { # setup matplotlib to use latex for output
"pgf.texsystem": "pdflatex", # change this if using xetex or lautex
"text.usetex": True, # use LaTeX to write all text
"font.family": "serif",
"font.serif": [], # blank entries should cause plots to inherit fonts from the document
"font.sans-serif": [],
"font.monospace": [],
"axes.labelsize": 10, # LaTeX default is 10pt font.
"font.size": 8,
"legend.fontsize": 7, # Make the legend/label fonts a little smaller
"xtick.labelsize": 7,
"ytick.labelsize": 7,
"pgf.preamble": [
r"\usepackage[utf8x]{inputenc}", # use utf8 fonts becasue your computer can handle it :)
r"\usepackage[T1]{fontenc}", # plots will be generated using this preamble
r"\usepackage{siunitx}",
r"\DeclareSIUnit[number-unit-product = {}] ",
r"\LSB{LSB}",
]
}
mpl.rcParams.update(pgf_with_latex)
import matplotlib.pyplot as pl
import numpy as np
fig=pl.figure(figsize=(3,2))
ax1 = fig.add_subplot(111)
dat=np.random.normal(-120-60,40,200000).astype(int)
bins=np.arange(int(np.amin(dat))-.5,127.5,2)
ax1.hist(dat, bins = bins, stacked = True)
ax1.set_title("\\emph{(a)} minimal example")
ax1.set_yscale("log", nonposy="clip")
ax1.set_ylim(0.8, 20000)
ax1.set_xlim(None, 130)
ax1.set_ylabel("frequency")
ax1.set_xlabel("data")
ax1.set_xticks([-300,-200, -127,0,127])
fig.tight_layout(h_pad=1,w_pad=0.2)
pl.savefig('test.png', bbox_inches='tight',dpi=600)
pl.savefig('test.pdf', bbox_inches='tight',dpi=600)
Вывод вышеуказанного кода: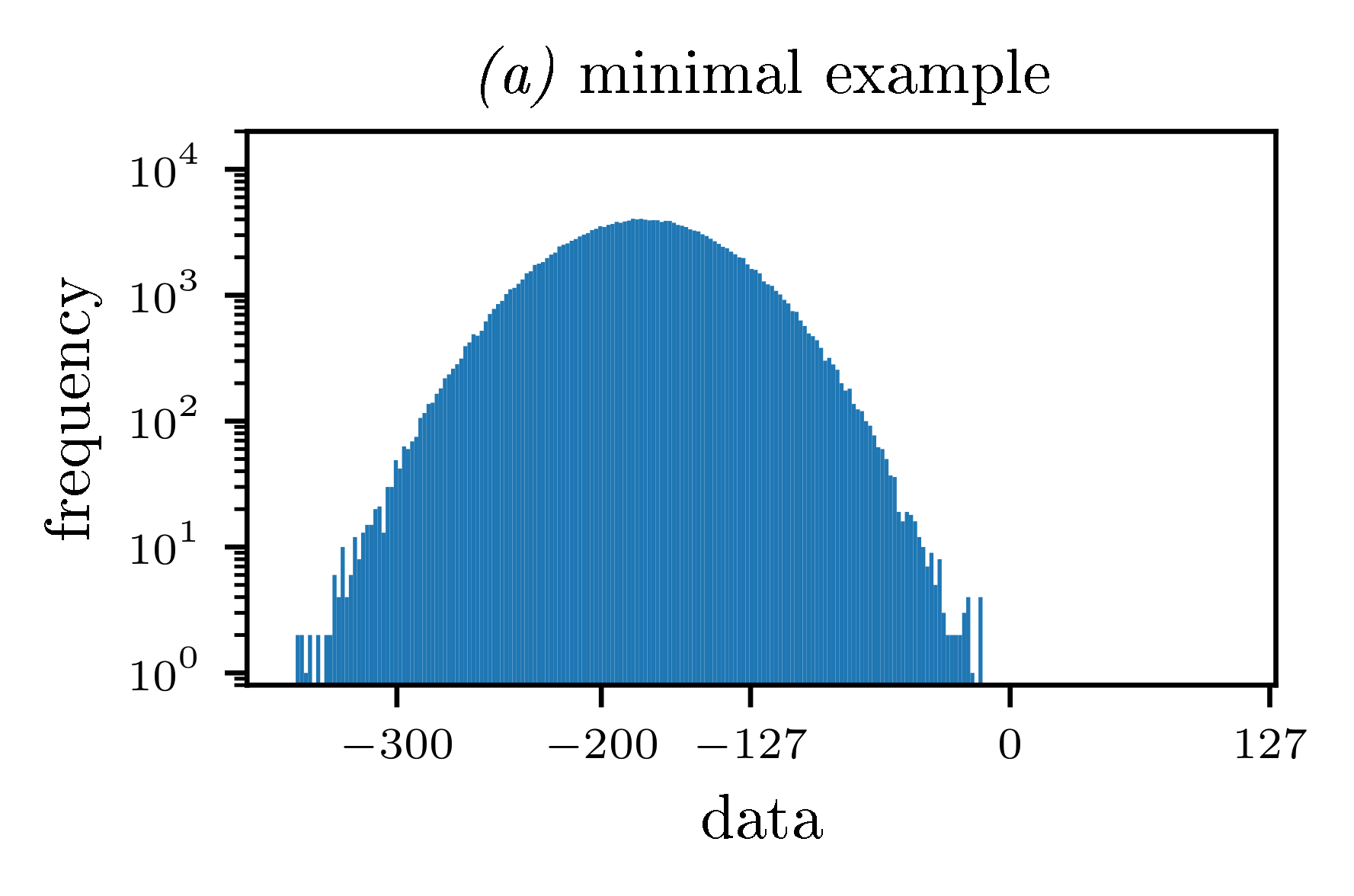
1 ответ
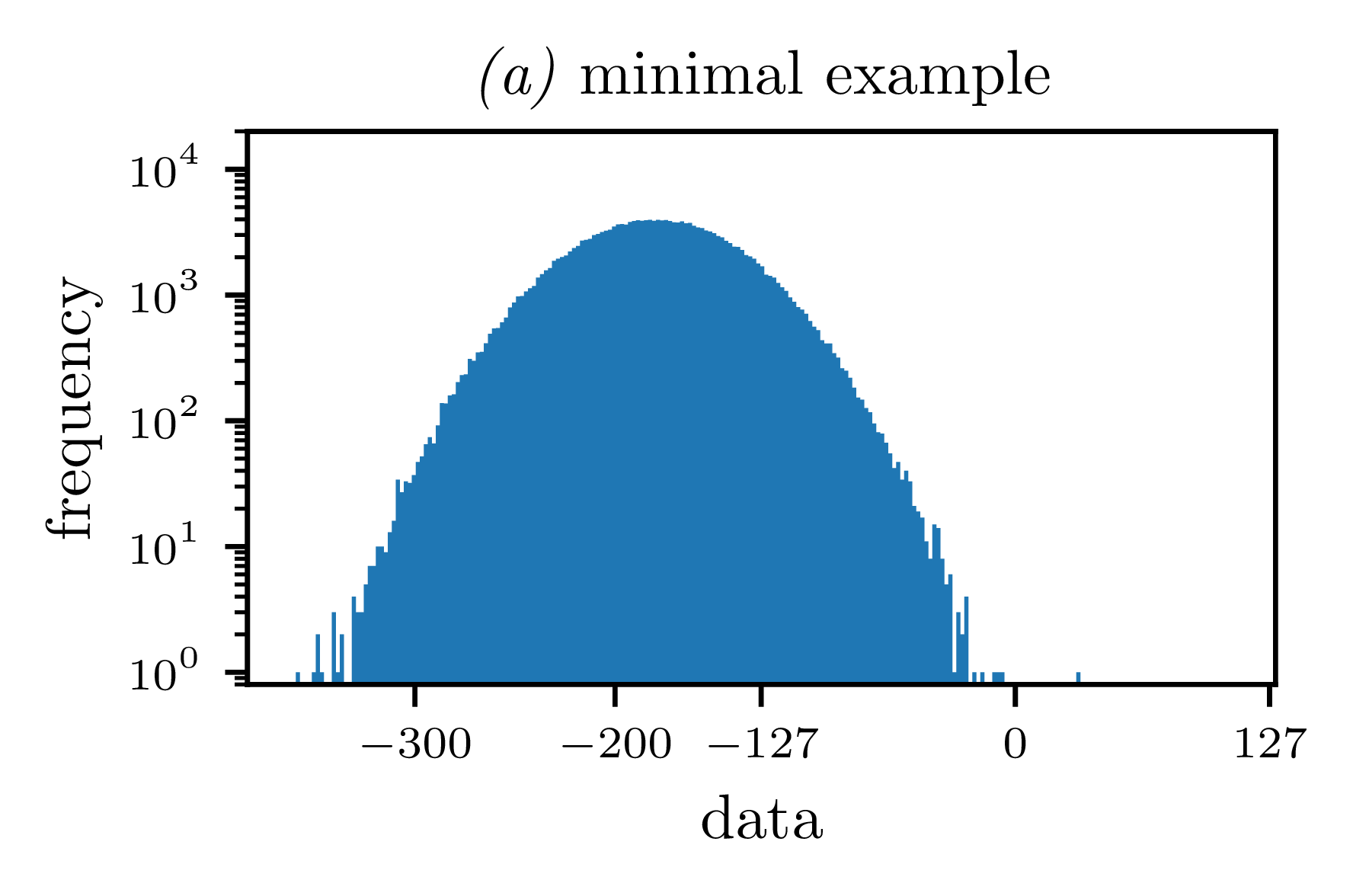
1. Не использовать pgf backend
Как отметил @unutbu в своем (к сожалению, теперь удаленном) ответе, отсутствие использования бэкэнда pgf фактически приведет к ожидаемому графику.
Удаление линии
mpl.use('pgf')
дам
2. Шаг функции
Если по какой-то причине нельзя избежать использования pgf backend, можно обойти это путем использования функции шага для построения гистограммы. Удаление ax1.hist(...) из кода и замены его на
hist, ex = np.histogram(dat, bins = bins)
ax1.fill_between(bins[:-1], hist, lw=0.0, step="post")
дает