networkD3 не рисует ожидаемую интерактивную сеть
У меня есть следующие данные:
links <- read.table(header = T, as.is = T, text = '
from to type
p13 p1 hyperlink
p13 p2 hyperlink
p13 p3 hyperlink
p13 p4 hyperlink
p13 p5 hyperlink
p13 p11 hyperlink
p12 p10 hyperlink
p12 p8 hyperlink
p12 p9 hyperlink
p14 p6 hyperlink
p14 p7 hyperlink
')
nodes <- read.table(header = T, as.is = T, text = '
id name node.type
p1 Pi Protein
p2 PPi Protein
p3 SAM Protein
p4 L-Met Protein
p5 H2O Protein
p6 SAH Protein
p7 Ade-Rib Protein
p8 LHCYS Protein
p9 tetrahydropteroyltri-L-glutamate Protein
p10 5-methyltetrahydropteroyltriglutamate-homocysteine Protein
p11 ATP Protein
p12 reaction1 Reaction
p13 reaction2 Reaction
p14 reaction3 Reaction
')
Я использую пакет networkd3, у меня есть следующий код:
library("networkD3")
links.d3 <- data.frame(from=as.numeric(factor(links$from))-1,
to=as.numeric(factor(links$to))-1 )
nodes.d3 <- cbind(idn=factor(nodes$name, levels=nodes$name), nodes)
forceNetwork(Links = links.d3,
Nodes = nodes.d3,
Source="from",
Target="to",
NodeID = "name",
Group = "node.type",
linkWidth = 1,
linkColour = "#afafaf",
fontSize=12,
zoom=T,
colourScale = JS(ColourScale),
legend=T,
opacity = 0.8,
charge=-300,
arrows = FALSE,
bounded = TRUE,
opacityNoHover = 2,
width = NULL,
height = NULL,
clickAction = myClick)
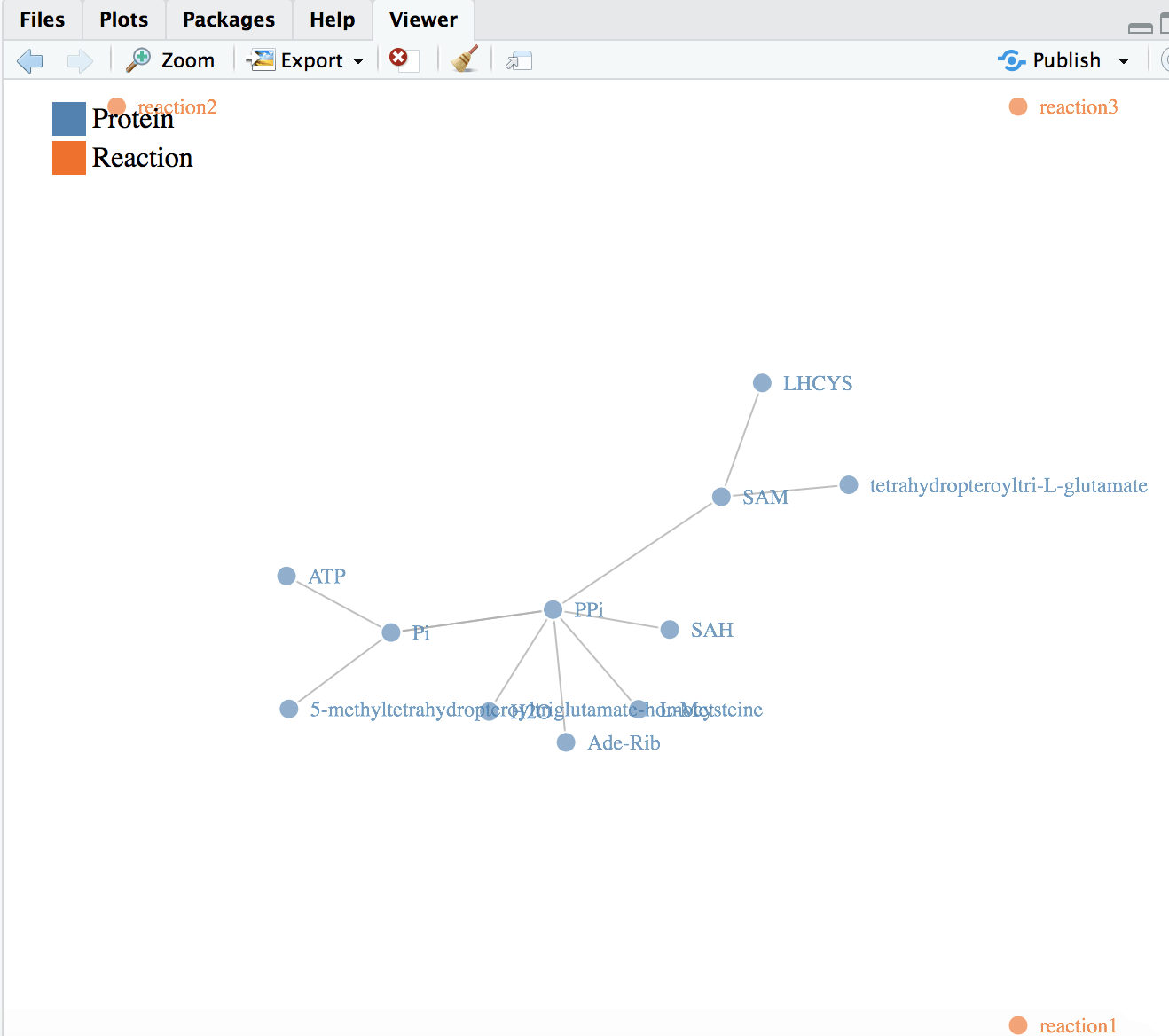
p12, p13 и p14 являются реакциями. Ожидаемый результат заключается в том, что каждый белок должен подключаться к какой-либо реакции, кроме H2O, L-Met и LHCYS, эти три должны подключаться к двум отдельным реакциям.
Вот что я получаю:
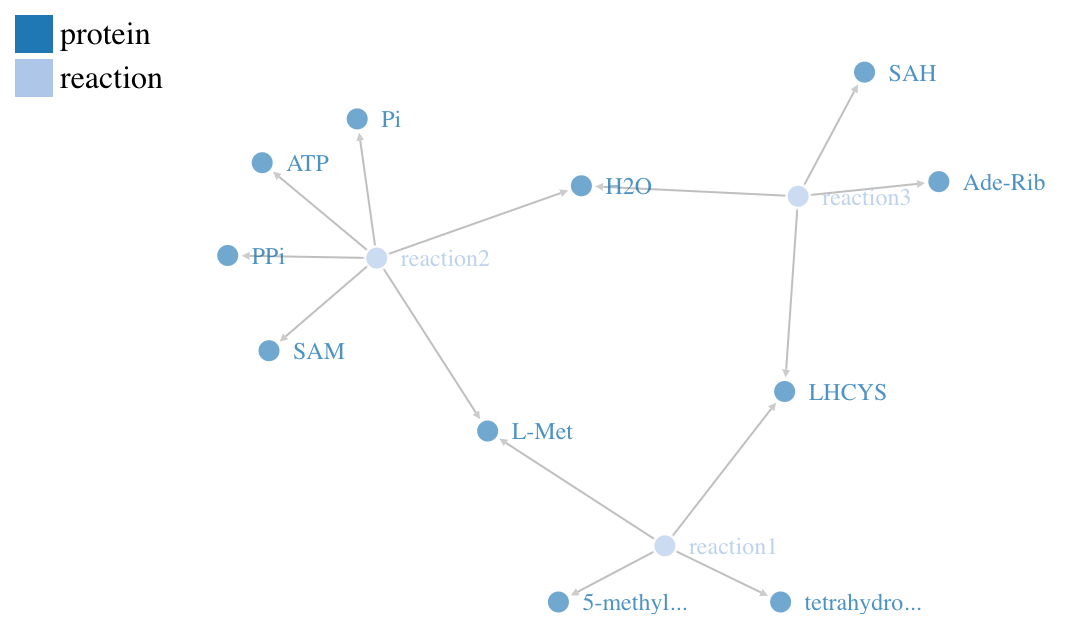
Приведите пример того, что я ожидаю:
Может кто-нибудь объяснить, почему и достичь желаемого результата?
1 ответ
Решение
Во-первых, вам не хватает ссылок в ваших ссылочных данных, которые определяют, что вы хотите показать:
- p12 -> p4 (реакция 1 -> L-Met)
- p14 -> p5 (реакция3 -> H2O)
- p14 -> p8 (реакция3 -> LHCYS)
Во-вторых, я не понимаю, что вы пытались сделать, создавая links.d3 data.frame, но from а также to переменные / столбцы должны быть индексом узла в nodes data.frame, который вы можете создать, как показано ниже...
links <- read.table(header = T, as.is = T, text = '
from to type
p13 p1 hyperlink
p13 p2 hyperlink
p13 p3 hyperlink
p13 p4 hyperlink
p13 p5 hyperlink
p13 p11 hyperlink
p12 p10 hyperlink
p12 p8 hyperlink
p12 p9 hyperlink
p12 p4 hyperlink
p14 p6 hyperlink
p14 p7 hyperlink
p14 p5 hyperlink
p14 p8 hyperlink
')
nodes <- read.table(header = T, as.is = T, text = '
id name node.type
p1 Pi Protein
p2 PPi Protein
p3 SAM Protein
p4 L-Met Protein
p5 H2O Protein
p6 SAH Protein
p7 Ade-Rib Protein
p8 LHCYS Protein
p9 tetrahydropteroyltri-L-glutamate Protein
p10 5-methyltetrahydropteroyltriglutamate-homocysteine Protein
p11 ATP Protein
p12 reaction1 Reaction
p13 reaction2 Reaction
p14 reaction3 Reaction
')
links.d3 <- data.frame(from = match(links$from, nodes$id) - 1,
to = match(links$to, nodes$id) - 1,
value = 1)
library("networkD3")
forceNetwork(Links = links.d3, Nodes = nodes, Source = "from", Target = "to",
Value = "value", NodeID = "name", Group = "node.type",
linkColour = "#afafaf", fontSize = 12, zoom = T, legend = T,
opacity = 0.8, charge = -300, arrows = TRUE, bounded = TRUE,
opacityNoHover = 2)