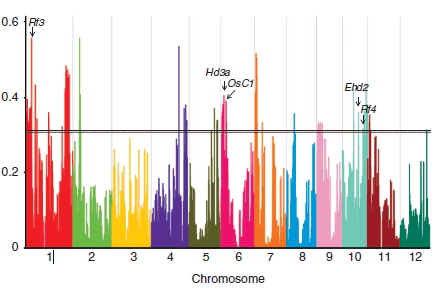
Как я могу указать группы (например, хромосомы) на непрерывной оси, используя ggplot2?
Это то, что я хочу построить:
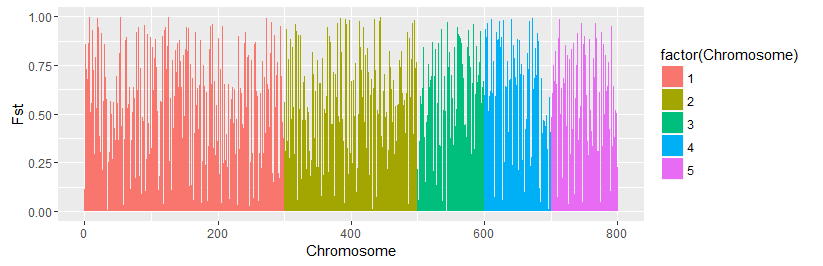
Используя g gplot2, я могу построить правильные столбцы, но с номером локуса, указанным на оси x вместо номера хромосомы. Он просто отображает номер каждого столбца, например 1,2,...,500, но не показывает хромосому. Как мне этого добиться?
Воспроизводимый пример:
p <- ggplot(data = info, aes(x = number,y= Fst,fill = Chromesome))
p + geom_bar(stat = "identity") + labs(x = "Chromosome", y =Fst)
number Chromosome Fst
1 1 0.5
2 1 0.6
3 2 0.7
4 2 0.9
5 3 0.7
6 3 0.1
7 4 0.3
8 4 0.4
9 5 0.5
10 5 0.6
11 6 0.7
12 6 0.8
13 7 0.9
1 ответ
Очевидным способом было бы использовать scale_x_continuous и установите ручные перерывы и метки, однако...
Один из способов, который я использовал ранее, - это использовать фасеты со свободными масштабами и пространством.
Создать случайные данные
info <- data.frame(number = 1:800,
Fst = runif(800),
Chromosome = rep(1:5, c(300, 200, 100, 100, 100)))
Ваш сюжет
Обратите внимание, что у вас были некоторые орфографические ошибки, которые привели к ошибкам.
p <- ggplot(data = info, aes(x = number, y = Fst, fill = factor(Chromosome))) +
geom_bar(stat = "identity") + labs(x = "Chromosome", y = "Fst")
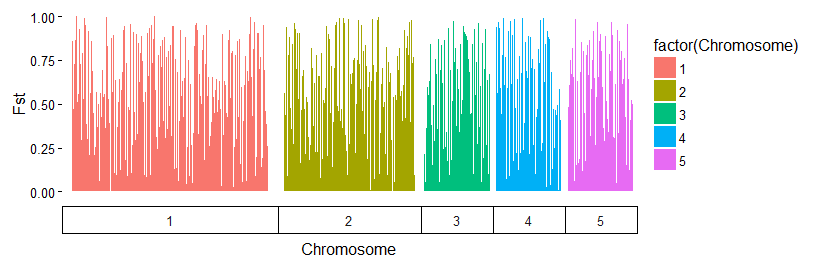
Добавить фасеты и настроить тему
p + facet_grid(~Chromosome, scales = 'free_x', space = 'free_x', switch = 'x') +
theme_classic() +
theme(axis.text.x = element_blank(),
axis.ticks.x = element_blank(),
panel.margin = unit(0, "lines"))
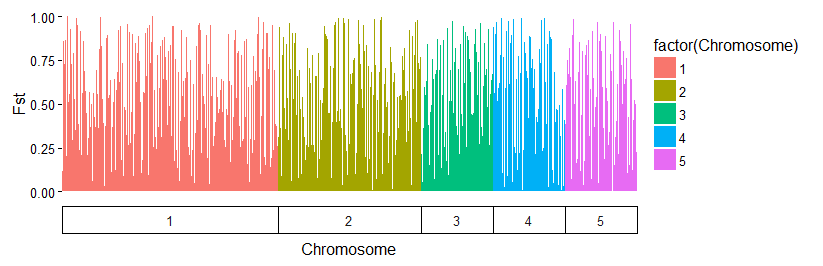
Вы можете отключить расширение оси, чтобы столбцы были точно рядом друг с другом, но лично мне очень нравятся промежутки.
p + facet_grid(~Chromosome, scales = 'free_x', space = 'free_x', switch = 'x') +
theme_classic() +
theme(axis.text.x = element_blank(),
axis.ticks.x = element_blank(),
panel.margin = unit(0, "lines")) +
scale_x_continuous(expand = c(0, 0))