Numpy: декартово произведение точек массива x и y на один массив точек 2D
У меня есть два массива, которые определяют оси X и Y сетки. Например:
x = numpy.array([1,2,3])
y = numpy.array([4,5])
Я хотел бы сгенерировать декартово произведение этих массивов для генерации:
array([[1,4],[2,4],[3,4],[1,5],[2,5],[3,5]])
В некотором смысле это не очень неэффективно, так как мне нужно делать это много раз в цикле. Я предполагаю, что преобразование их в список Python и использование itertools.product и возвращение к массиву не самая эффективная форма.
18 ответов
>>> numpy.transpose([numpy.tile(x, len(y)), numpy.repeat(y, len(x))])
array([[1, 4],
[2, 4],
[3, 4],
[1, 5],
[2, 5],
[3, 5]])
См. Использование numpy для создания массива всех комбинаций из двух массивов для общего решения для вычисления декартового произведения N массивов.
Канонический cartesian_product (почти)
Есть много подходов к этой проблеме с различными свойствами. Некоторые из них быстрее, чем другие, а некоторые более общего назначения. После долгих испытаний и настроек я обнаружил, что следующая функция, которая вычисляет n-мерную cartesian_product, быстрее, чем большинство других для многих входов. Для пары подходов, которые немного более сложны, но во многих случаях даже немного быстрее, см. Ответ Пола Панцера.
Учитывая этот ответ, это уже не самая быстрая реализация декартового произведения в numpy что я в курсе. Тем не менее, я думаю, что его простота будет продолжать делать его полезным ориентиром для будущих улучшений:
def cartesian_product(*arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
Стоит отметить, что эта функция использует ix_ необычным образом; в то время как документированное использование ix_ состоит в том, чтобы генерировать индексы в массиве, так получается, что массивы с одинаковой формой могут использоваться для широковещательного присваивания. Большое спасибо mgilson, который вдохновил меня попробовать ix_ таким образом, и unutbu, который предоставил несколько чрезвычайно полезных отзывов об этом ответе, включая предложение использовать numpy.result_type,
Известные альтернативы
Иногда быстрее записать непрерывные блоки памяти в порядке Фортрана. Это основа этой альтернативы, cartesian_product_transpose, который оказался быстрее на некоторых аппаратных средствах, чем cartesian_product (увидеть ниже). Однако ответ Пола Панцера, использующий тот же принцип, еще быстрее. Тем не менее, я включаю это здесь для заинтересованных читателей:
def cartesian_product_transpose(*arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
Поняв подход Panzer, я написал новую версию, которая почти такая же быстрая, как и его, и такая же простая, как cartesian_product:
def cartesian_product_simple_transpose(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([la] + [len(a) for a in arrays], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[i, ...] = a
return arr.reshape(la, -1).T
Похоже, что это имеет некоторые постоянные накладные расходы, что делает его медленным, чем Panzer, для небольших входов. Но для больших входных данных во всех тестах, которые я запускал, он работает так же хорошо, как и его самая быстрая реализация (cartesian_product_transpose_pp).
В следующих разделах я включаю некоторые тесты других альтернатив. Теперь они несколько устарели, но вместо дублирующих усилий я решил оставить их здесь вне исторического интереса. Актуальные тесты см. В ответе Panzer, а также в ответе Нико Шлёмера.
Тесты против альтернатив
Вот набор тестов, которые показывают повышение производительности, которое предоставляют некоторые из этих функций относительно ряда альтернатив. Все тесты, показанные здесь, были выполнены на четырехъядерном компьютере под управлением Mac OS 10.12.5, Python 3.6.1 и numpy 1.12.1. Известно, что различия в аппаратном и программном обеспечении дают разные результаты, поэтому YMMV. Запустите эти тесты для себя, чтобы быть уверенным!
Определения:
import numpy
import itertools
from functools import reduce
### Two-dimensional products ###
def repeat_product(x, y):
return numpy.transpose([numpy.tile(x, len(y)),
numpy.repeat(y, len(x))])
def dstack_product(x, y):
return numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
### Generalized N-dimensional products ###
def cartesian_product(*arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(*arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
# from https://stackru.com/a/1235363/577088
def cartesian_product_recursive(*arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:,0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m,1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m,1:] = out[0:m,1:]
return out
def cartesian_product_itertools(*arrays):
return numpy.array(list(itertools.product(*arrays)))
### Test code ###
name_func = [('repeat_product',
repeat_product),
('dstack_product',
dstack_product),
('cartesian_product',
cartesian_product),
('cartesian_product_transpose',
cartesian_product_transpose),
('cartesian_product_recursive',
cartesian_product_recursive),
('cartesian_product_itertools',
cartesian_product_itertools)]
def test(in_arrays, test_funcs):
global func
global arrays
arrays = in_arrays
for name, func in test_funcs:
print('{}:'.format(name))
%timeit func(*arrays)
def test_all(*in_arrays):
test(in_arrays, name_func)
# `cartesian_product_recursive` throws an
# unexpected error when used on more than
# two input arrays, so for now I've removed
# it from these tests.
def test_cartesian(*in_arrays):
test(in_arrays, name_func[2:4] + name_func[-1:])
x10 = [numpy.arange(10)]
x50 = [numpy.arange(50)]
x100 = [numpy.arange(100)]
x500 = [numpy.arange(500)]
x1000 = [numpy.arange(1000)]
Результаты теста:
In [2]: test_all(*(x100 * 2))
repeat_product:
67.5 µs ± 633 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
dstack_product:
67.7 µs ± 1.09 µs per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product:
33.4 µs ± 558 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product_transpose:
67.7 µs ± 932 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
cartesian_product_recursive:
215 µs ± 6.01 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_itertools:
3.65 ms ± 38.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
In [3]: test_all(*(x500 * 2))
repeat_product:
1.31 ms ± 9.28 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
dstack_product:
1.27 ms ± 7.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product:
375 µs ± 4.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_transpose:
488 µs ± 8.88 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
cartesian_product_recursive:
2.21 ms ± 38.4 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
105 ms ± 1.17 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [4]: test_all(*(x1000 * 2))
repeat_product:
10.2 ms ± 132 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
dstack_product:
12 ms ± 120 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product:
4.75 ms ± 57.1 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_transpose:
7.76 ms ± 52.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_recursive:
13 ms ± 209 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
422 ms ± 7.77 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Во всех случаях, cartesian_product как определено в начале этого ответа, самый быстрый.
Для тех функций, которые принимают произвольное количество входных массивов, стоит проверить производительность, когда len(arrays) > 2 также. (Пока я не могу определить, почему cartesian_product_recursive выдает ошибку в этом случае, я удалил ее из этих тестов.)
In [5]: test_cartesian(*(x100 * 3))
cartesian_product:
8.8 ms ± 138 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_transpose:
7.87 ms ± 91.5 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
518 ms ± 5.5 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [6]: test_cartesian(*(x50 * 4))
cartesian_product:
169 ms ± 5.1 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_transpose:
184 ms ± 4.32 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_itertools:
3.69 s ± 73.5 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [7]: test_cartesian(*(x10 * 6))
cartesian_product:
26.5 ms ± 449 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
cartesian_product_transpose:
16 ms ± 133 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
cartesian_product_itertools:
728 ms ± 16 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [8]: test_cartesian(*(x10 * 7))
cartesian_product:
650 ms ± 8.14 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
cartesian_product_transpose:
518 ms ± 7.09 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
cartesian_product_itertools:
8.13 s ± 122 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Как показывают эти тесты, cartesian_product остается конкурентоспособным, пока число входных массивов не превысит (примерно) четыре. После этого, cartesian_product_transpose имеет небольшое преимущество
Стоит повторить, что пользователи с другим оборудованием и операционными системами могут видеть разные результаты. Например, unutbu сообщает о следующих результатах этих тестов с использованием Ubuntu 14.04, Python 3.4.3 и numpy 1.14.0.dev0+b7050a9:
>>> %timeit cartesian_product_transpose(x500, y500)
1000 loops, best of 3: 682 µs per loop
>>> %timeit cartesian_product(x500, y500)
1000 loops, best of 3: 1.55 ms per loop
Ниже я подробно расскажу о предыдущих тестах, которые я проводил в этом направлении. Относительная производительность этих подходов со временем менялась для разных аппаратных средств и разных версий Python и numpy, Хотя это не сразу полезно для людей, использующих современные версии numpy, это иллюстрирует, как все изменилось с первой версии этого ответа.
Простая альтернатива: meshgrid + dstack
В настоящее время принятый ответ использует tile а также repeat транслировать два массива вместе. Но meshgrid Функция делает практически то же самое. Вот вывод tile а также repeat перед передачей для транспонирования:
In [1]: import numpy
In [2]: x = numpy.array([1,2,3])
...: y = numpy.array([4,5])
...:
In [3]: [numpy.tile(x, len(y)), numpy.repeat(y, len(x))]
Out[3]: [array([1, 2, 3, 1, 2, 3]), array([4, 4, 4, 5, 5, 5])]
И вот вывод meshgrid:
In [4]: numpy.meshgrid(x, y)
Out[4]:
[array([[1, 2, 3],
[1, 2, 3]]), array([[4, 4, 4],
[5, 5, 5]])]
Как видите, он практически идентичен. Нам нужно только изменить результат, чтобы получить точно такой же результат.
In [5]: xt, xr = numpy.meshgrid(x, y)
...: [xt.ravel(), xr.ravel()]
Out[5]: [array([1, 2, 3, 1, 2, 3]), array([4, 4, 4, 5, 5, 5])]
Вместо того, чтобы изменить форму на этом этапе, мы могли бы передать результат meshgrid в dstack и измените впоследствии, что экономит некоторую работу:
In [6]: numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
Out[6]:
array([[1, 4],
[2, 4],
[3, 4],
[1, 5],
[2, 5],
[3, 5]])
Вопреки утверждению в этом комментарии, я не видел никаких доказательств того, что различные входные данные будут давать выходные данные различной формы, и, как показано выше, они делают очень похожие вещи, поэтому было бы довольно странно, если бы они это сделали. Пожалуйста, дайте мне знать, если вы найдете контрпример.
тестирование meshgrid + dstack против repeat + transpose
Относительная эффективность этих двух подходов со временем изменилась. В более ранней версии Python (2.7) результат с использованием meshgrid + dstack был заметно быстрее для небольших входов. (Обратите внимание, что эти тесты взяты из старой версии этого ответа.) Определения:
>>> def repeat_product(x, y):
... return numpy.transpose([numpy.tile(x, len(y)),
numpy.repeat(y, len(x))])
...
>>> def dstack_product(x, y):
... return numpy.dstack(numpy.meshgrid(x, y)).reshape(-1, 2)
...
Для ввода среднего размера я увидел значительное ускорение. Но я повторил эти тесты с более поздними версиями Python (3.6.1) и numpy (1.12.1), на более новой машине. Два подхода сейчас практически идентичны.
Старый тест
>>> x, y = numpy.arange(500), numpy.arange(500)
>>> %timeit repeat_product(x, y)
10 loops, best of 3: 62 ms per loop
>>> %timeit dstack_product(x, y)
100 loops, best of 3: 12.2 ms per loop
Новый тест
In [7]: x, y = numpy.arange(500), numpy.arange(500)
In [8]: %timeit repeat_product(x, y)
1.32 ms ± 24.7 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
In [9]: %timeit dstack_product(x, y)
1.26 ms ± 8.47 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
Как всегда, YMMV, но это говорит о том, что в последних версиях Python и numpy они взаимозаменяемы.
Обобщенные функции продукта
В целом, мы можем ожидать, что использование встроенных функций будет быстрее для небольших входов, в то время как для больших входов встроенная функция может быть быстрее. Кроме того, для обобщенного n-мерного произведения, tile а также repeat не поможет, потому что у них нет четких многомерных аналогов. Так что стоит исследовать поведение специализированных функций.
Большинство соответствующих тестов появляются в начале этого ответа, но вот некоторые из тестов, выполненных на более ранних версиях Python и numpy для сравнения.
cartesian функция, определенная в другом ответе, используется для довольно хорошего выполнения при больших входах (Это то же самое, что и вызываемая функция cartesian_product_recursive выше.) Для того, чтобы сравнить cartesian в dstack_prodct Мы используем только два измерения.
Здесь снова старый тест показал значительную разницу, в то время как новый тест почти не показал.
Старый тест
>>> x, y = numpy.arange(1000), numpy.arange(1000)
>>> %timeit cartesian([x, y])
10 loops, best of 3: 25.4 ms per loop
>>> %timeit dstack_product(x, y)
10 loops, best of 3: 66.6 ms per loop
Новый тест
In [10]: x, y = numpy.arange(1000), numpy.arange(1000)
In [11]: %timeit cartesian([x, y])
12.1 ms ± 199 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
In [12]: %timeit dstack_product(x, y)
12.7 ms ± 334 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
Как прежде, dstack_product все еще бьет cartesian в меньших масштабах.
Новый тест (старый избыточный тест не показан)
In [13]: x, y = numpy.arange(100), numpy.arange(100)
In [14]: %timeit cartesian([x, y])
215 µs ± 4.75 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
In [15]: %timeit dstack_product(x, y)
65.7 µs ± 1.15 µs per loop (mean ± std. dev. of 7 runs, 10000 loops each)
Эти различия, я думаю, интересны и заслуживают записи; но они академичны в конце концов. Как показали тесты в начале этого ответа, все эти версии почти всегда работают медленнее, чем cartesian_product определено в самом начале этого ответа - что само по себе немного медленнее, чем самые быстрые реализации среди ответов на этот вопрос.
Вы можете просто сделать обычное понимание списка в Python
x = numpy.array([1,2,3])
y = numpy.array([4,5])
[[x0, y0] for x0 in x for y0 in y]
который должен дать вам
[[1, 4], [1, 5], [2, 4], [2, 5], [3, 4], [3, 5]]
Я также заинтересовался этим и провел небольшое сравнение производительности, возможно, несколько понятнее, чем в ответе @senderle.
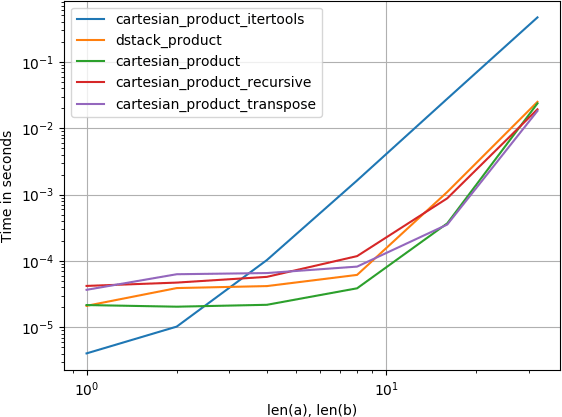
Для двух массивов (классический случай):
Для четырех массивов:
(Обратите внимание, что длина массивов здесь составляет всего несколько десятков записей.)
Код для воспроизведения сюжетов:
from functools import reduce
import itertools
import numpy
import perfplot
def dstack_product(arrays):
return numpy.dstack(
numpy.meshgrid(*arrays, indexing='ij')
).reshape(-1, len(arrays))
# Generalized N-dimensional products
def cartesian_product(arrays):
la = len(arrays)
dtype = numpy.find_common_type([a.dtype for a in arrays], [])
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[..., i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = reduce(numpy.multiply, broadcasted[0].shape), len(broadcasted)
dtype = numpy.find_common_type([a.dtype for a in arrays], [])
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
# from https://stackru.com/a/1235363/577088
def cartesian_product_recursive(arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:, 0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m, 1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m, 1:] = out[0:m, 1:]
return out
def cartesian_product_itertools(arrays):
return numpy.array(list(itertools.product(*arrays)))
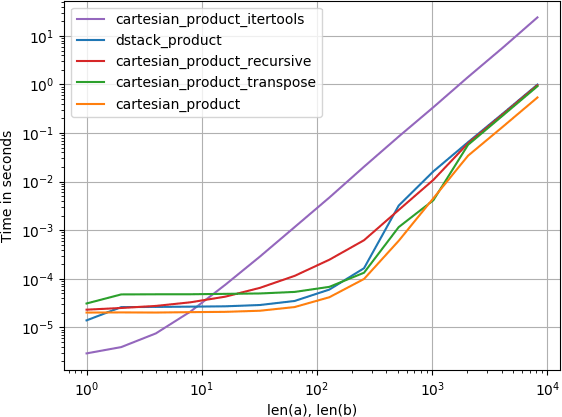
perfplot.show(
setup=lambda n: 4*(numpy.arange(n, dtype=float),),
n_range=[2**k for k in range(6)],
kernels=[
dstack_product,
cartesian_product,
cartesian_product_transpose,
cartesian_product_recursive,
cartesian_product_itertools
],
logx=True,
logy=True,
xlabel='len(a), len(b)',
equality_check=None
)
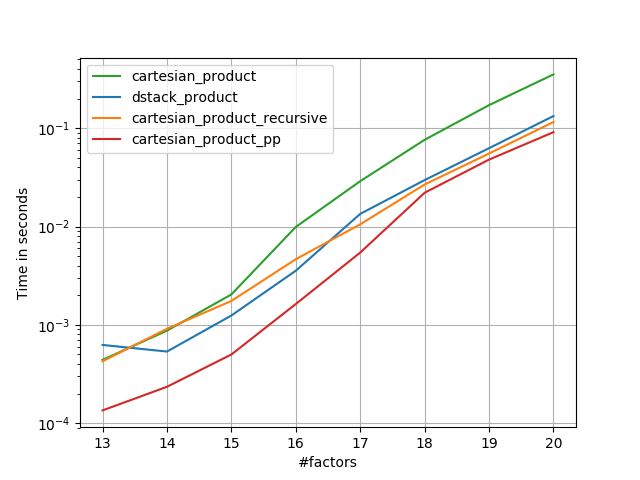
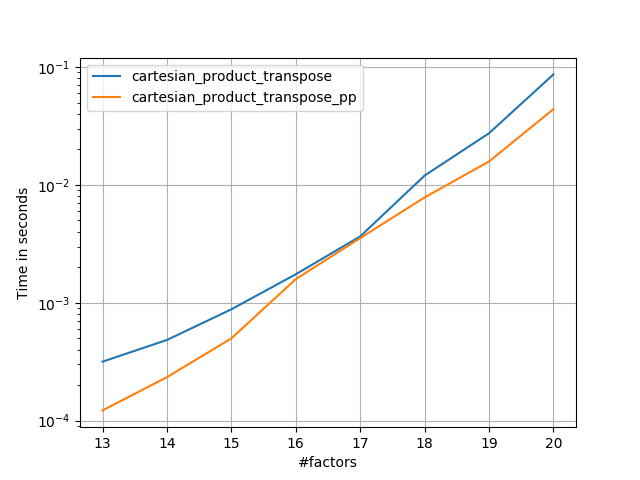
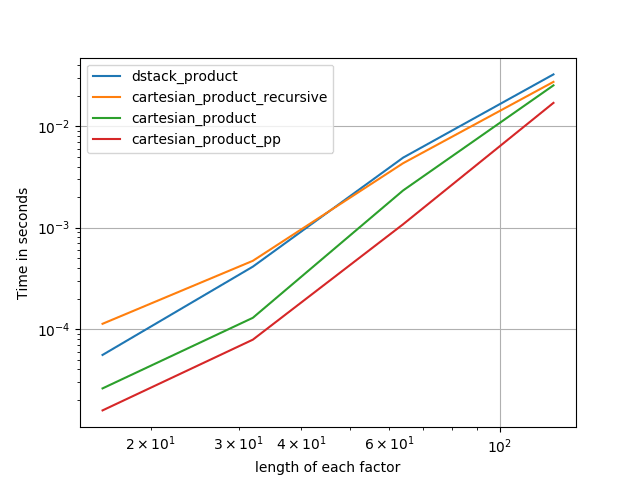
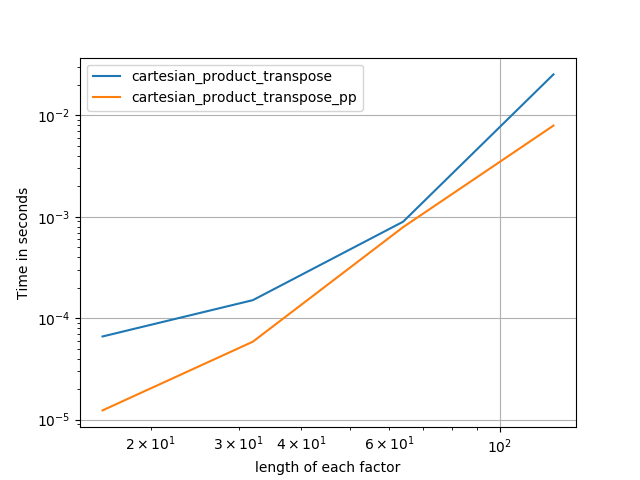
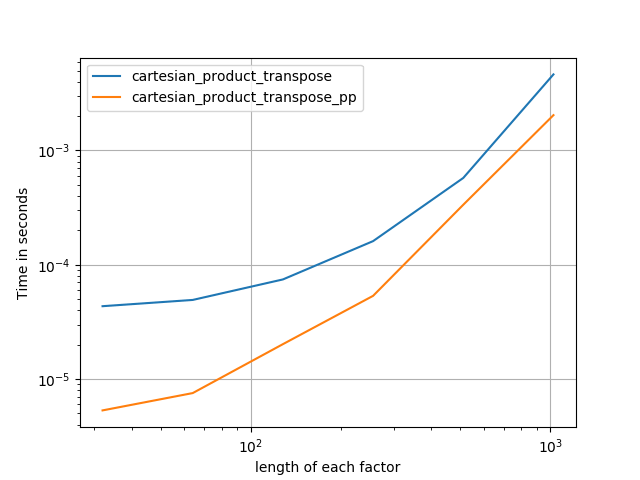
Основываясь на образцовой работе @senderle, я придумал две версии - одну для C и одну для разметки Fortran - которые часто бывают немного быстрее.
cartesian_product_transpose_ppесть - в отличие от @senderle'scartesian_product_transposeкоторый использует совсем другую стратегию - версиюcartesion_productэто использует более благоприятное расположение памяти транспонирования + некоторые очень незначительные оптимизации.cartesian_product_ppпридерживается оригинального расположения памяти. Что делает его быстрым, так это использование непрерывного копирования. Непрерывные копии оказываются настолько быстрыми, что копирование полного блока памяти, даже если только часть его содержит действительные данные, предпочтительнее, чем копирование действительных битов.
Несколько перфлотов. Я сделал отдельные для макетов C и Fortran, потому что это разные задачи IMO.
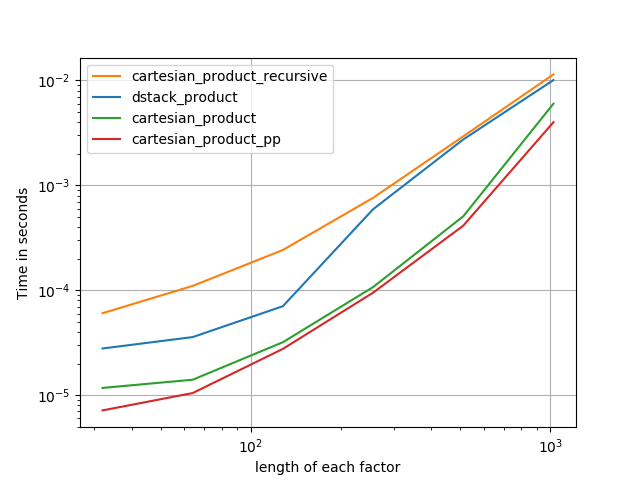
Имена, оканчивающиеся на 'pp' - мои подходы.
1) много крошечных факторов (2 элемента каждый)
2) много мелких факторов (4 элемента каждый)
3) три фактора равной длины
4) два фактора равной длины
Код (нужно делать отдельные прогоны для каждого сюжета, потому что я не могу понять, как выполнить сброс; также нужно соответствующим образом редактировать / комментировать / выводить):
import numpy
import numpy as np
from functools import reduce
import itertools
import timeit
import perfplot
def dstack_product(arrays):
return numpy.dstack(
numpy.meshgrid(*arrays, indexing='ij')
).reshape(-1, len(arrays))
def cartesian_product_transpose_pp(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty((la, *map(len, arrays)), dtype=dtype)
idx = slice(None), *itertools.repeat(None, la)
for i, a in enumerate(arrays):
arr[i, ...] = a[idx[:la-i]]
return arr.reshape(la, -1).T
def cartesian_product(arrays):
la = len(arrays)
dtype = numpy.result_type(*arrays)
arr = numpy.empty([len(a) for a in arrays] + [la], dtype=dtype)
for i, a in enumerate(numpy.ix_(*arrays)):
arr[...,i] = a
return arr.reshape(-1, la)
def cartesian_product_transpose(arrays):
broadcastable = numpy.ix_(*arrays)
broadcasted = numpy.broadcast_arrays(*broadcastable)
rows, cols = numpy.prod(broadcasted[0].shape), len(broadcasted)
dtype = numpy.result_type(*arrays)
out = numpy.empty(rows * cols, dtype=dtype)
start, end = 0, rows
for a in broadcasted:
out[start:end] = a.reshape(-1)
start, end = end, end + rows
return out.reshape(cols, rows).T
from itertools import accumulate, repeat, chain
def cartesian_product_pp(arrays, out=None):
la = len(arrays)
L = *map(len, arrays), la
dtype = numpy.result_type(*arrays)
arr = numpy.empty(L, dtype=dtype)
arrs = *accumulate(chain((arr,), repeat(0, la-1)), np.ndarray.__getitem__),
idx = slice(None), *itertools.repeat(None, la-1)
for i in range(la-1, 0, -1):
arrs[i][..., i] = arrays[i][idx[:la-i]]
arrs[i-1][1:] = arrs[i]
arr[..., 0] = arrays[0][idx]
return arr.reshape(-1, la)
def cartesian_product_itertools(arrays):
return numpy.array(list(itertools.product(*arrays)))
# from https://stackru.com/a/1235363/577088
def cartesian_product_recursive(arrays, out=None):
arrays = [numpy.asarray(x) for x in arrays]
dtype = arrays[0].dtype
n = numpy.prod([x.size for x in arrays])
if out is None:
out = numpy.zeros([n, len(arrays)], dtype=dtype)
m = n // arrays[0].size
out[:, 0] = numpy.repeat(arrays[0], m)
if arrays[1:]:
cartesian_product_recursive(arrays[1:], out=out[0:m, 1:])
for j in range(1, arrays[0].size):
out[j*m:(j+1)*m, 1:] = out[0:m, 1:]
return out
### Test code ###
if False:
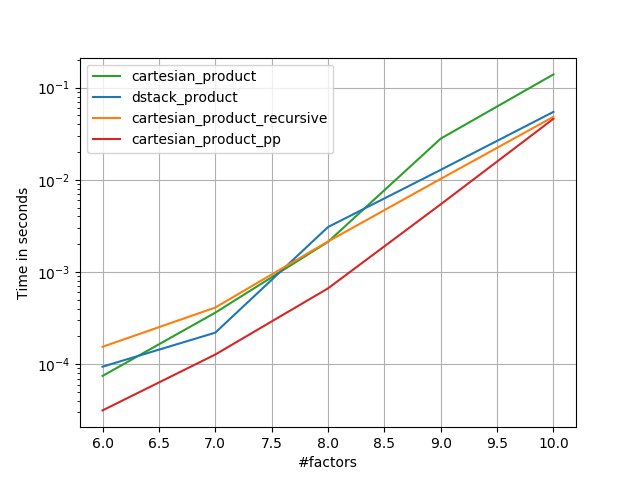
perfplot.save('cp_4el_high.png',
setup=lambda n: n*(numpy.arange(4, dtype=float),),
n_range=list(range(6, 11)),
kernels=[
dstack_product,
cartesian_product_recursive,
cartesian_product,
# cartesian_product_transpose,
cartesian_product_pp,
# cartesian_product_transpose_pp,
],
logx=False,
logy=True,
xlabel='#factors',
equality_check=None
)
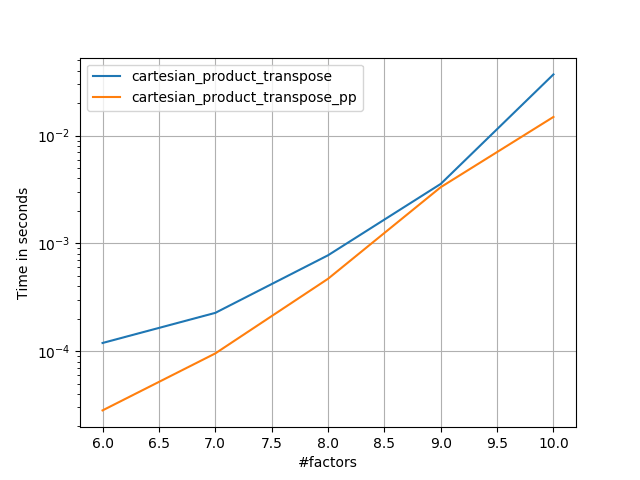
else:
perfplot.save('cp_2f_T.png',
setup=lambda n: 2*(numpy.arange(n, dtype=float),),
n_range=[2**k for k in range(5, 11)],
kernels=[
# dstack_product,
# cartesian_product_recursive,
# cartesian_product,
cartesian_product_transpose,
# cartesian_product_pp,
cartesian_product_transpose_pp,
],
logx=True,
logy=True,
xlabel='length of each factor',
equality_check=None
)
По состоянию на октябрь 2017 года у numpy теперь есть общий np.stack функция, которая принимает параметр оси. Используя его, мы можем получить "обобщенное декартово произведение", используя технику "dstack and meshgrid":
import numpy as np
def cartesian_product(*arrays):
ndim = len(arrays)
return np.stack(np.meshgrid(*arrays), axis=-1).reshape(-1, ndim)
Обратите внимание на axis=-1 параметр. Это последняя (самая внутренняя) ось в результате. Это эквивалентно использованию axis=ndim,
Еще один комментарий: поскольку декартовы произведения взрываются очень быстро, если нам не нужно по какой-то причине реализовать массив в памяти, если продукт очень большой, мы можем захотеть использовать itertools и используйте значения на лету.
Пакет Scikit-learn имеет быструю реализацию именно этого:
from sklearn.utils.extmath import cartesian
product = cartesian((x,y))
Обратите внимание, что соглашение этой реализации отличается от того, что вы хотите, если вы заботитесь о порядке вывода. Для вашего точного заказа вы можете сделать
product = cartesian((y,x))[:, ::-1]
Некоторое время я использовал ответ @kennytm, но при попытке сделать то же самое в TensorFlow, но обнаружил, что TensorFlow не имеет эквивалента numpy.repeat(), После небольшого эксперимента, я думаю, я нашел более общее решение для произвольных векторов точек.
Для NumPy:
import numpy as np
def cartesian_product(*args: np.ndarray) -> np.ndarray:
"""
Produce the cartesian product of arbitrary length vectors.
Parameters
----------
np.ndarray args
vector of points of interest in each dimension
Returns
-------
np.ndarray
the cartesian product of size [m x n] wherein:
m = prod([len(a) for a in args])
n = len(args)
"""
for i, a in enumerate(args):
assert a.ndim == 1, "arg {:d} is not rank 1".format(i)
return np.concatenate([np.reshape(xi, [-1, 1]) for xi in np.meshgrid(*args)], axis=1)
и для TensorFlow:
import tensorflow as tf
def cartesian_product(*args: tf.Tensor) -> tf.Tensor:
"""
Produce the cartesian product of arbitrary length vectors.
Parameters
----------
tf.Tensor args
vector of points of interest in each dimension
Returns
-------
tf.Tensor
the cartesian product of size [m x n] wherein:
m = prod([len(a) for a in args])
n = len(args)
"""
for i, a in enumerate(args):
tf.assert_rank(a, 1, message="arg {:d} is not rank 1".format(i))
return tf.concat([tf.reshape(xi, [-1, 1]) for xi in tf.meshgrid(*args)], axis=1)
В более общем случае, если у вас есть два двумерных числовых массива a и b, и вы хотите объединить каждую строку a с каждой строкой b (декартово произведение строк, вроде соединения в базе данных), вы можете использовать этот метод:
import numpy
def join_2d(a, b):
assert a.dtype == b.dtype
a_part = numpy.tile(a, (len(b), 1))
b_part = numpy.repeat(b, len(a), axis=0)
return numpy.hstack((a_part, b_part))
Это также легко сделать с помощью метода itertools.product.
from itertools import product
import numpy as np
x = np.array([1, 2, 3])
y = np.array([4, 5])
cart_prod = np.array(list(product(*[x, y])),dtype='int32')
Результат: массив ([
[1, 4],
[1, 5],
[2, 4],
[2, 5],
[3, 4],
[3, 5]], dtype = int32)
Время выполнения: 0,000155 с
Самое быстрое, что вы можете получить, это либо объединить выражение генератора с функцией map:
import numpy
import datetime
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = (item for sublist in [list(map(lambda x: (x,i),a)) for i in b] for item in sublist)
print (list(foo))
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Выходы (фактически весь итоговый список печатается):
[(0, 0), (1, 0), ...,(998, 199), (999, 199)]
execution time: 1.253567 s
или с помощью выражения двойного генератора:
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = ((x,y) for x in a for y in b)
print (list(foo))
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Выходы (весь список напечатан):
[(0, 0), (1, 0), ...,(998, 199), (999, 199)]
execution time: 1.187415 s
Учтите, что большая часть времени вычислений уходит на команду печати. Расчеты генератора в остальном прилично эффективны. Без печати время расчета составляет:
execution time: 0.079208 s
для выражения генератора + функция карты и:
execution time: 0.007093 s
для выражения двойного генератора.
Если на самом деле вы хотите вычислить фактическое произведение каждой из пар координат, самое быстрое - это решить его как произведение матрицы на кусочки:
a = np.arange(1000)
b = np.arange(200)
start = datetime.datetime.now()
foo = np.dot(np.asmatrix([[i,0] for i in a]), np.asmatrix([[i,0] for i in b]).T)
print (foo)
print ('execution time: {} s'.format((datetime.datetime.now() - start).total_seconds()))
Выходы:
[[ 0 0 0 ..., 0 0 0]
[ 0 1 2 ..., 197 198 199]
[ 0 2 4 ..., 394 396 398]
...,
[ 0 997 1994 ..., 196409 197406 198403]
[ 0 998 1996 ..., 196606 197604 198602]
[ 0 999 1998 ..., 196803 197802 198801]]
execution time: 0.003869 s
и без печати (в этом случае это не сильно экономит, так как на самом деле распечатывается только крошечный кусочек матрицы):
execution time: 0.003083 s
В конкретном случае, когда вам нужно выполнить простые операции, такие как сложение для каждой пары, вы можете ввести дополнительное измерение и позволить вещанию делать свою работу:
>>> a, b = np.array([1,2,3]), np.array([10,20,30])
>>> a[None,:] + b[:,None]
array([[11, 12, 13],
[21, 22, 23],
[31, 32, 33]])
Я не уверен, есть ли подобный способ на самом деле получить сами пары.
Я немного опоздал на вечеринку, но воспользовался хитрым вариантом этой проблемы. Скажем, мне нужно декартово произведение нескольких массивов, но это декартово произведение оказывается намного больше, чем память компьютера (однако вычисления, выполняемые с этим продуктом, бывают быстрыми или, по крайней мере, распараллеливаемыми).
Очевидное решение состоит в том, чтобы разделить этот декартово произведение на куски и обрабатывать эти куски один за другим (как бы "поточно"). Вы можете легко сделать это с помощьюitertools.product, но это ужасно медленно. Кроме того, ни одно из предложенных здесь решений (таких быстрых) не дает нам такой возможности. Предлагаемое мной решение использует Numba и немного быстрее, чем "каноническое". cartesian_product упоминается здесь. Это довольно долго, потому что я пытался оптимизировать его везде, где мог.
import numba as nb
import numpy as np
from typing import List
@nb.njit(nb.types.Tuple((nb.int32[:, :],
nb.int32[:]))(nb.int32[:],
nb.int32[:],
nb.int64, nb.int64))
def cproduct(sizes: np.ndarray, current_tuple: np.ndarray, start_idx: int, end_idx: int):
"""Generates ids tuples from start_id to end_id"""
assert len(sizes) >= 2
assert start_idx < end_idx
tuples = np.zeros((end_idx - start_idx, len(sizes)), dtype=np.int32)
tuple_idx = 0
# stores the current combination
current_tuple = current_tuple.copy()
while tuple_idx < end_idx - start_idx:
tuples[tuple_idx] = current_tuple
current_tuple[0] += 1
# using a condition here instead of including this in the inner loop
# to gain a bit of speed: this is going to be tested each iteration,
# and starting a loop to have it end right away is a bit silly
if current_tuple[0] == sizes[0]:
# the reset to 0 and subsequent increment amount to carrying
# the number to the higher "power"
current_tuple[0] = 0
current_tuple[1] += 1
for i in range(1, len(sizes) - 1):
if current_tuple[i] == sizes[i]:
# same as before, but in a loop, since this is going
# to get called less often
current_tuple[i + 1] += 1
current_tuple[i] = 0
else:
break
tuple_idx += 1
return tuples, current_tuple
def chunked_cartesian_product_ids(sizes: List[int], chunk_size: int):
"""Just generates chunks of the cartesian product of the ids of each
input arrays (thus, we just need their sizes here, not the actual arrays)"""
prod = np.prod(sizes)
# putting the largest number at the front to more efficiently make use
# of the cproduct numba function
sizes = np.array(sizes, dtype=np.int32)
sorted_idx = np.argsort(sizes)[::-1]
sizes = sizes[sorted_idx]
if chunk_size > prod:
chunk_bounds = (np.array([0, prod])).astype(np.int64)
else:
num_chunks = np.maximum(np.ceil(prod / chunk_size), 2).astype(np.int32)
chunk_bounds = (np.arange(num_chunks + 1) * chunk_size).astype(np.int64)
chunk_bounds[-1] = prod
current_tuple = np.zeros(len(sizes), dtype=np.int32)
for start_idx, end_idx in zip(chunk_bounds[:-1], chunk_bounds[1:]):
tuples, current_tuple = cproduct(sizes, current_tuple, start_idx, end_idx)
# re-arrange columns to match the original order of the sizes list
# before yielding
yield tuples[:, np.argsort(sorted_idx)]
def chunked_cartesian_product(*arrays, chunk_size=2 ** 25):
"""Returns chunks of the full cartesian product, with arrays of shape
(chunk_size, n_arrays). The last chunk will obviously have the size of the
remainder"""
array_lengths = [len(array) for array in arrays]
for array_ids_chunk in chunked_cartesian_product_ids(array_lengths, chunk_size):
slices_lists = [arrays[i][array_ids_chunk[:, i]] for i in range(len(arrays))]
yield np.vstack(slices_lists).swapaxes(0,1)
def cartesian_product(*arrays):
"""Actual cartesian product, not chunked, still fast"""
total_prod = np.prod([len(array) for array in arrays])
return next(chunked_cartesian_product(*arrays, total_prod))
a = np.arange(0, 3)
b = np.arange(8, 10)
c = np.arange(13, 16)
for cartesian_tuples in chunked_cartesian_product(*[a, b, c], chunk_size=5):
print(cartesian_tuples)
Это выведет наш декартово произведение кусками по 5 троек:
[[ 0 8 13]
[ 0 8 14]
[ 0 8 15]
[ 1 8 13]
[ 1 8 14]]
[[ 1 8 15]
[ 2 8 13]
[ 2 8 14]
[ 2 8 15]
[ 0 9 13]]
[[ 0 9 14]
[ 0 9 15]
[ 1 9 13]
[ 1 9 14]
[ 1 9 15]]
[[ 2 9 13]
[ 2 9 14]
[ 2 9 15]]
Если вы хотите понять, что здесь делается, интуиция, стоящая за njitted Функция состоит в том, чтобы перечислить каждое "число" в причудливой числовой базе, элементы которой будут состоять из размеров входных массивов (вместо того же числа в обычных двоичных, десятичных или шестнадцатеричных основаниях).
Очевидно, что такое решение интересно для крупногабаритных изделий. Для маленьких накладные расходы могут быть немного дорогими.
ПРИМЕЧАНИЕ: поскольку numba все еще находится в стадии интенсивной разработки, я использую numba 0.50 для его запуска с python 3.6.
Это обобщенная версия принятого ответа (декартово произведение нескольких массивов с использованиемnumpy.tileиnumpy.repeatфункции).
from functors import reduce
from operator import mul
def cartesian_product(arrays):
return np.vstack(
np.tile(
np.repeat(arrays[j], reduce(mul, map(len, arrays[j+1:]), 1)),
reduce(mul, map(len, arrays[:j]), 1),
)
for j in range(len(arrays))
).T
Обратите внимание: если вам нужно декартово произведение целых диапазонов, вы можете использовать следующие функции:
numpy.indices((100, 200)).reshape(2, -1).T
или
numpy.mgrid[0:100, 0:200].reshape(2, -1).T
оба вернутся
array([[ 0, 0],
[ 0, 1],
[ 0, 2],
...,
[ 99, 197],
[ 99, 198],
[ 99, 199]])
numpy.indicesварьируется только от 0 до каждого из указанных размеров, но это намного быстрее. Обе функции также могут работать более чем в двух измерениях, например:
numpy.indices((100, 200, 300)).reshape(3, -1).T
и
numpy.mgrid[0:100, 0:200, 0:300].reshape(3, -1).T
Вдохновленный ответом Ашкана , вы также можете попробовать следующее.
>>> x, y = np.meshgrid(x, y)
>>> np.concatenate([x.flatten().reshape(-1,1), y.flatten().reshape(-1,1)], axis=1)
Это даст вам необходимое декартово произведение!
Еще один:
>>>x1, y1 = np.meshgrid(x, y)
>>>np.c_[x1.ravel(), y1.ravel()]
array([[1, 4],
[2, 4],
[3, 4],
[1, 5],
[2, 5],
[3, 5]])
Если вы хотите использовать PyTorch, я думаю, что это очень эффективно:
>>> import torch
>>> torch.cartesian_prod(torch.as_tensor(x), torch.as_tensor(y))
tensor([[1, 4],
[1, 5],
[2, 4],
[2, 5],
[3, 4],
[3, 5]])
и вы можете легко получитьnumpyмножество:
>>> torch.cartesian_prod(torch.as_tensor(x), torch.as_tensor(y)).numpy()
array([[1, 4],
[1, 5],
[2, 4],
[2, 5],
[3, 4],
[3, 5]])