Эффективная фильтрация вблизи / внутри кластеров после их обнаружения - python
По сути я применил DBSCAN алгоритм (sklearn) с евклидовым расстоянием на подмножестве моих исходных данных. Я нашел свои кластеры, и все в порядке: за исключением того факта, что я хочу сохранить только те значения, которые достаточно далеки от тех, на которых я не проводил анализ. У меня есть новая дистанция для тестирования такого нового материала, и я хотел понять, как это сделать БЕЗ многочисленных вложенных циклов.
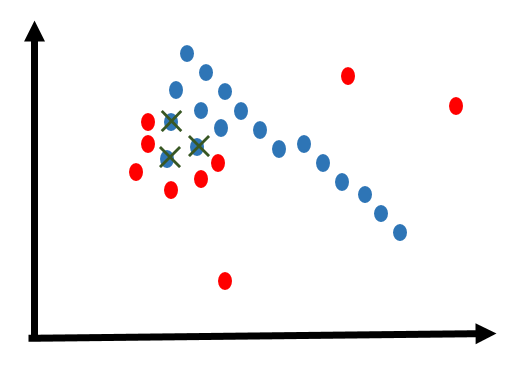
на картинке:
мои найденные кластеры выделены синим цветом, тогда как красные - это точки, к которым я не хочу приближаться. кресты - это точки, принадлежащие скоплению, которые вырезаются в пределах нового расстояния, которое я указал.
теперь, сколько я мог сделать что-то в этом роде:
for i in red_points:
for j in blu_points:
if dist(i,j) < given_dist:
original_dataframe.remove(j)
Я отказываюсь верить, что нет векторизованного метода. Кроме того, я не могу позволить себе сделать так, как описано выше, просто потому, что у меня будут огромные таблицы для работы, и я бы хотел избежать испарения моего процессора.
любые предложения приветствуются
2 ответа
Конечно, вы можете векторизовать это, но тогда оно все равно будет O(n*m). Лучшие алгоритмы поиска соседей не векторизованы. например, kd-tree и ball-tree.
Оба доступны в sklearn и используются модулем DBSCAN. Пожалуйста, смотрите sklearn.neighbors пакет.
Если вам нужны точные ответы, самой быстрой реализацией должен быть калькулятор парных расстояний sklearn: http://scikit-learn.org/stable/modules/generated/sklearn.metrics.pairwise.pairwise_distances.html
Если вы можете принять приблизительный ответ, вы можете сделать это лучше с помощью queryradius() дерева kd: http://scikit-learn.org/stable/modules/generated/sklearn.neighbors.KDTree.html