Объединение пересекающихся векторов в списке в R
У меня есть список векторов следующим образом.
data <- list(v1=c("a", "b", "c"), v2=c("g", "h", "k"),
v3=c("c", "d"), v4=c("n", "a"), v5=c("h", "i"))
Я пытаюсь добиться следующего
1) Проверьте, пересекаются ли какие-либо векторы.
2) Если пересекающиеся векторы найдены, найдите их объединение.
Таким образом, желаемый результат
out <- list(v1=c("a", "b", "c", "d", "n"), v2=c("g", "h", "k", "i"))
Я могу получить объединение группы пересекающихся множеств следующим образом.
Reduce(union, list(data[[1]], data[[3]], data[[4]]))
Reduce(union, list(data[[2]], data[[5]])
Как сначала определить пересекающиеся векторы? Есть ли способ разделить список на списки групп пересекающихся векторов?
Обновить
Вот попытка использования data.table. Получает желаемые результаты. Но все еще медленно для больших списков, как в этом примере набора данных.
datasets.
data <- sapply(data, function(x) paste(x, collapse=", "))
data <- as.data.frame(data, stringsAsFactors = F)
repeat {
M <- nrow(data)
data <- data.table( data , key = "data" )
data <- data[ , list(dataelement = unique(unlist(strsplit(data , ", " )))), by = list(data)]
data <- data.table(data , key = "dataelement" )
data <- data[, list(data = paste0(sort(unique(unlist(strsplit(data, split=", ")))), collapse=", ")), by = "dataelement"]
data$dataelement <- NULL
data <- unique(data)
N <- nrow(data)
if (M == N)
break
}
data <- strsplit(as.character(data$data) , "," )
6 ответов
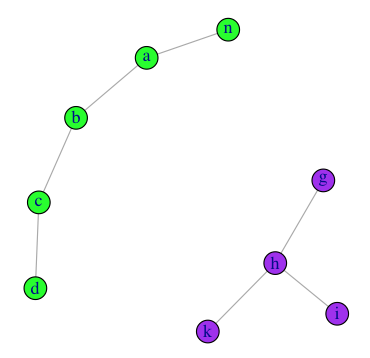
Это похоже на проблему с графиком, поэтому мне нравится использовать igraph библиотека для этого, используя ваши данные образца, вы можете сделать
library(igraph)
#build edgelist
el <- do.call("rbind",lapply(data, embed, 2))
#make a graph
gg <- graph.edgelist(el, directed=F)
#partition the graph into disjoint sets
split(V(gg)$name, clusters(gg)$membership)
# $`1`
# [1] "b" "a" "c" "d" "n"
#
# $`2`
# [1] "h" "g" "k" "i"
И мы можем просмотреть результаты с
V(gg)$color=c("green","purple")[clusters(gg)$membership]
plot(gg)
Вот еще один подход с использованием только базы R
Обновить
Следующее обновление после комментария Акруна и с его примерами данных:
data <- list(v1=c('g', 'k'), v2= letters[1:4], v3= c('b', 'c', 'd', 'a'))
Модифицированная функция:
x <- lapply(seq_along(data), function(i) {
if(!any(data[[i]] %in% unlist(data[-i]))) {
data[[i]]
} else if (any(data[[i]] %in% unlist(data[seq_len(i-1)]))) {
NULL
} else {
z <- lapply(data[-seq_len(i)], intersect, data[[i]])
z <- names(z[sapply(z, length) >= 1L])
if (is.null(z)) NULL else union(data[[i]], unlist(data[z]))
}
})
x[!sapply(x, is.null)]
#[[1]]
#[1] "g" "k"
#
#[[2]]
#[1] "a" "b" "c" "d"
Это хорошо работает с исходными образцами данных, образцами данных MrFlick и образцами данных akrun.
Будь проклята эффективность, а вы, люди, вообще спите? Только база R и намного медленнее, чем самый быстрый ответ. Так как я написал это, мог бы также опубликовать это.
f.union = function(x) {
repeat{
n = length(x)
m = matrix(F, nrow = n, ncol = n)
for (i in 1:n){
for (j in 1:n) {
m[i,j] = any(x[[i]] %in% x[[j]])
}
}
o = apply(m, 2, function(v) Reduce(union, x[v]))
if (all(apply(m, 1, sum)==1)) {return(o)} else {x=unique(o)}
}
}
f.union(data)
[[1]]
[1] "a" "b" "c" "d" "n"
[[2]]
[1] "g" "h" "k" "i"
Потому что мне нравится быть медленным. (загруженная библиотека вне эталона)
Unit: microseconds
expr min lq mean median uq max neval
vlo() 896.435 1070.6540 1315.8194 1129.4710 1328.6630 7859.999 1000
akrun() 596.263 658.6590 789.9889 694.1360 804.9035 3470.158 1000
flick() 805.854 928.8160 1160.9509 1001.8345 1172.0965 5780.824 1000
josh() 2427.752 2693.0065 3344.8671 2943.7860 3524.1550 16505.909 1000 <- deleted :-(
doc() 254.462 288.9875 354.6084 302.6415 338.9565 2734.795 1000
Один вариант будет использовать combn а затем найти пересечения. Там были бы более легкие варианты.
indx <- combn(names(data),2)
lst <- lapply(split(indx, col(indx)),
function(i) Reduce(`intersect`,data[i]))
indx1 <- names(lst[sapply(lst, length)>0])
indx2 <- indx[,as.numeric(indx1)]
indx3 <- apply(indx2,2, sort)
lapply(split(1:ncol(indx3), indx3[1,]),
function(i) unique(unlist(data[c(indx3[,i])], use.names=FALSE)))
#$v1
#[1] "a" "b" "c" "d" "n"
#$v2
#[1] "g" "h" "k" "i"
Обновить
Вы могли бы использовать combnPrim от library(gRbase) чтобы сделать это еще быстрее. Использование немного большего набора данных
library(gRbase)
set.seed(25)
data <- setNames(lapply(1:1e3,function(i)sample(letters,
sample(1:20), replace=FALSE)), paste0("v", 1:1000))
и сравнивая с fastest, Это модифицированные функции, основанные на комментариях OP к @docendo discimus.
akrun2M <- function(){
ind <- sapply(seq_along(data), function(i){#copied from @docendo discimus
!any(data[[i]] %in% unlist(data[-i]))
})
data1 <- data[!ind]
indx <- combnPrim(names(data1),2)
lst <- lapply(split(indx, col(indx)),
function(i) Reduce(`intersect`,data1[i]))
indx1 <- names(lst[sapply(lst, length)>0])
indx2 <- indx[,as.numeric(indx1)]
indx3 <- apply(indx2,2, sort)
c(data[ind],lapply(split(1:ncol(indx3), indx3[1,]),
function(i) unique(unlist(data[c(indx3[,i])], use.names=FALSE))))
}
doc2 <- function(){
x <- lapply(seq_along(data), function(i) {
if(!any(data[[i]] %in% unlist(data[-i]))) {
data[[i]]
}
else {
z <- unlist(data[names(unlist(lapply(data[-c(1:i)],
intersect, data[[i]])))])
if (is.null(z)){
z
}
else union(data[[i]], z)
}
})
x[!sapply(x, is.null)]
}
Ориентиры
microbenchmark(doc2(), akrun2M(), times=10L)
# Unit: seconds
# expr min lq mean median uq max neval cld
# doc2() 35.43687 53.76418 54.77813 54.34668 62.86665 67.76754 10 b
#akrun2M() 26.64997 28.74721 38.02259 35.35081 47.56781 49.82158 10 a
Я столкнулся с похожей проблемой, которая побудила меня искать решение повсюду. Я, наконец, нашел очень хороший вариант благодаря ряду замечательных участников, однако, когда я увидел этот пост, я подумал, что напишу свою собственную функцию для этой цели. На самом деле это не изящно и не слишком медленно, но я думаю, что это довольно эффективно и пока что может помочь, пока я не внесу некоторые улучшения:
anoush <- function(x) {
stopifnot(is.list(x))
ind <- lapply(1:length(x), function(a) {
vec <- c()
for(i in 1:length(x)) {
if(length(unique(base::intersect(x[[a]], x[[i]]))) > 0) {
vec <- c(vec, i)
}
}
vec
})
dup_ind <- lapply(1:length(ind), function(a) {
out <- c()
for(i in 1:length(ind)) {
if(length(unique(base::intersect(ind[[a]], ind[[i]]))) > 0) {
out[[i]] <- union(ind[[a]], ind[[i]])
}
vec2 <- Reduce("union", out)
}
vec2
})
un <- unlist(dup_ind)
res <- Map(`[`, dup_ind, relist(!duplicated(un), skeleton = dup_ind))
res2 <- Filter(length, res)
sapply(res2, function(a) unique(unlist(lapply(a, function(b) `[[`(x, b)))))
}
Образец данных OP
> anoush(data)
[[1]]
[1] "a" "b" "c" "d" "n"
[[2]]
[1] "g" "h" "k" "i"
Уважаемый @ akrun образец данных
data <- list(v1=c('g', 'k'), v2= letters[1:4], v3= c('b', 'c', 'd', 'a'))
> anoush(data)
[[1]]
[1] "g" "k"
[[2]]
[1] "a" "b" "c" "d"
В общем, вы не можете сделать намного лучше / быстрее, чем алгоритм Флойда-Варшалла, который выглядит следующим образом:
library(Rcpp)
cppFunction(
"LogicalMatrix floyd(LogicalMatrix w){
int n = w.nrow();
for( int k = 0; k < n; k++ )
for( int i = 0; i < (n-1); i++ )
for( int j = i+1; j < n; j++ )
if( w(i,k) && w(k,j) ) {
w(i,j) = true;
w(j,i) = true;
}
return w;
}")
fw.union<-function(x) {
n<-length(x)
w<-matrix(F,nrow=n,ncol=n)
for( i in 1:n ) {
w[i,i]<-T
}
for( i in 1:(n-1) ) {
for( j in (i+1):n ) {
w[i,j]<-w[j,i]<- any(x[[i]] %in% x[[j]])
}
}
apply( unique( floyd(w) ), 1, function(y) { Reduce(union,x[y]) } )
}
Запуск тестов будет интересно, хотя. Предварительные тесты показывают, что моя реализация примерно в 2-3 раза быстрее, чем у Вло.