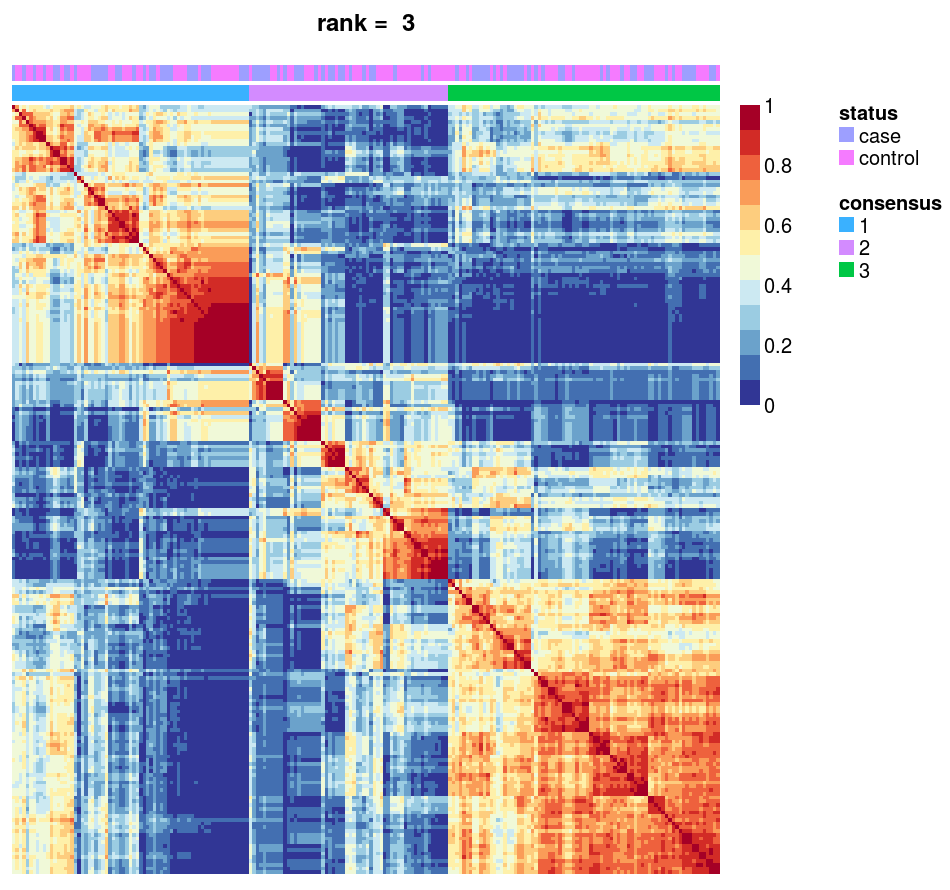
R NMF пакет: Как извлечь классификацию образцов?
В R-пакете NMF можно использовать consensusmap() для визуализации результатов. Графики показывают, какие образцы принадлежат к каким кластерам в "консенсусном" треке.
Я хотел бы извлечь этот пример классификации так, чтобы я получил кадр данных, как это:
Sample Cluster
S1 1
S2 1
S3 2
S4 1
. .
. .
S100 2
В пакете ConsensusClusterPlus это легко. Вы просто вытаскиваете результаты $consensusClass. Я не могу найти аналогичное решение для NMF-пакета. Я попытался взглянуть на необработанные данные графика, но они слишком запутаны, чтобы извлечь из них какой-либо смысл.
Вот иллюстрация проблемы: мне нужно выяснить, какой "статус" находится в пределах какого "консенсуса".
2 ответа
Прогуляться по всему дереву и сосчитать?
> v <- syntheticNMF(20, 3, 10)
> xx<-consensusmap(x)
> str(xx)
List of 4
$ Rowv : ..--[dendrogram w/ 2 branches and 10 members at h = 1, midpoint = 5.97, value = 3.4]
.. |--[dendrogram w/ 2 branches and 7 members at h = 1, midpoint = 3.69, value = 2.5]
.. | |--[dendrogram w/ 2 branches and 4 members at h = 0, midpoint = 2.12, value = 1.6]
.. | | |--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 1.25, value = 1.2]
.. | | | |--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.8]
.. | | | | |--leaf "2" ( value.2 = 0.4 )
.. | | | | `--leaf "1" ( value.1 = 0.4 )
.. | | | `--leaf "3" ( value.3 = 0.4 )
.. | | `--leaf "4" ( value.4 = 0.4 )
.. | `--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 1.25, value = 0.9]
.. | |--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.6]
.. | | |--leaf "6" ( value.6 = 0.3 )
.. | | `--leaf "5" ( value.5 = 0.3 )
.. | `--leaf "7" ( value.7 = 0.3 )
.. `--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 1.25, value = 0.9]
.. |--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.6]
.. | |--leaf "9" ( value.9 = 0.3 )
.. | `--leaf "8" ( value.8 = 0.3 )
.. `--leaf "10" ( value.10 = 0.3 )
$ rowInd: int [1:10] 2 1 3 4 6 5 7 9 8 10
$ Colv : ..--[dendrogram w/ 2 branches and 10 members at h = 1, midpoint = 3.03, value = 3.4]
.. |--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 0.75, value = 0.9]
.. | |--leaf "10" ( value.10 = 0.3 )
.. | `--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.6]
.. | |--leaf "8" ( value.8 = 0.3 )
.. | `--leaf "9" ( value.9 = 0.3 )
.. `--[dendrogram w/ 2 branches and 7 members at h = 1, midpoint = 2.31, value = 2.5]
.. |--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 0.75, value = 0.9]
.. | |--leaf "7" ( value.7 = 0.3 )
.. | `--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.6]
.. | |--leaf "5" ( value.5 = 0.3 )
.. | `--leaf "6" ( value.6 = 0.3 )
.. `--[dendrogram w/ 2 branches and 4 members at h = 0, midpoint = 0.875, value = 1.6]
.. |--leaf "4" ( value.4 = 0.4 )
.. `--[dendrogram w/ 2 branches and 3 members at h = 0, midpoint = 0.75, value = 1.2]
.. |--leaf "3" ( value.3 = 0.4 )
.. `--[dendrogram w/ 2 branches and 2 members at h = 0, midpoint = 0.5, value = 0.8]
.. |--leaf "1" ( value.1 = 0.4 )
.. `--leaf "2" ( value.2 = 0.4 )
$ colInd: int [1:10] 10 8 9 7 5 6 4 3 1 2
> lapply(cut(xx$Rowv,0.5)$lower, function(l) rapply(l, function(i) i))
[[1]]
[1] 2 1 3 4
[[2]]
[1] 6 5 7
[[3]]
[1] 9 8 10
NMF::predict(NMFfitX_object, what = "consensus")
Если у вас есть объект класса NMF.rank, вы можете получить доступ к содержащимся объектам NMFfitX для ранга = 3 с помощью:
NMF.rank_object$fit$`3`