R: аккуратная агрегация данных последовательности и визуализация пошаговых функций
У меня есть некоторые данные о пациентах, где отдельные пациенты меняют группы лечения с течением времени. Моя цель - визуализировать последовательность изменений группы и объединить эти данные в "профиль последовательности" для каждой группы лечения.
Для каждой группы лечения я хотел бы показать, когда это обычно происходит в цикле лечения (скажем, скорее в начале или в конце). Чтобы учесть различную длину последовательности, я хотел бы стандартизировать эти профили между n 0 (самое начало) и 1 (конец).
Я хотел бы найти эффективную подготовку данных и визуализацию.
Мининмал Пример
Структура данных
library(dplyr)
library(purrr)
library(ggplot2)
# minimal data
cj_df_raw <- tibble::tribble(
~id, ~group,
1, "A",
1, "B",
2, "A",
2, "B",
2, "A"
)
# compute "intervals" for each person [start, end]
cj_df_raw %>%
group_by(id) %>%
mutate(pos = row_number(),
len = length(id),
start = (pos - 1) / len,
end = pos / len) %>%
filter(group == "A")
#> # A tibble: 3 x 6
#> # Groups: id [2]
#> id group pos len start end
#> <dbl> <chr> <int> <int> <dbl> <dbl>
#> 1 1 A 1 2 0 0.5
#> 2 2 A 1 3 0 0.333
#> 3 2 A 3 3 0.667 1
(Таким образом, Id 1 был в группе A в первых 50% их последовательности, а Id 2 был в группе A в первых 33% и последних 33% их последовательности. Это означает, что 2 идентификатора находились между 0-33% последовательности, 1 между 33-50%, 0 между 50-66% и 1 выше 66%.)
Это результат, которого я хотел бы достичь, и я упускаю шанс эффективно преобразовать свои данные.
Желаемый результат
profile_treatmen_a <- tibble::tribble(
~x, ~y,
0, 0L,
0.33, 2L,
0.5, 1L,
0.66, 0L,
1, 1L,
1, 0L
)
profile_treatmen_a %>%
ggplot(aes(x, y)) +
geom_step(direction = "vh") +
expand_limits(x = c(0, 1), y = 0)
 (В идеале область под кривой должна быть затенена)
(В идеале область под кривой должна быть затенена)
Идеальное решение: через ggridges
Целью визуализации было бы сравнение "профиля последовательности" многих групп лечения одновременно. Если бы я мог подготовить данные соответствующим образом, я бы хотел использовать пакет ggridges для поразительного визуального сравнения групп лечения.
library(ggridges)
data.frame(group = rep(letters[1:2], each=20),
mean = rep(2, each=20)) %>%
mutate(count = runif(nrow(.))) %>%
ggplot(aes(x=count, y=group, fill=group)) +
geom_ridgeline(stat="binline", binwidth=0.5, scale=0.9)

2 ответа
Вы можете построить вспомогательные интервалы, а затем просто построить гистограмму. Поскольку каждый пациент находится в группе А или В, обе группы суммируют до 100%. С этими вспомогательными интервалами вы также можете легко переключаться на другие geoms,
library(tidyverse, warn.conflicts = FALSE)
library(ggplot2)
# create sample data
set.seed(42)
id <- 1:10 %>% map(~ rep(x = .x, times = runif(n = 1, min = 1, max = 6))) %>%
unlist()
group <- sample(x = c("A", "B"), size = length(id), replace = TRUE) %>%
as_factor()
df <- tibble(id, group)
glimpse(df)
#> Observations: 37
#> Variables: 2
#> $ id <int> 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 3, 3, 4, 4, 4, 4, 4, 5, 5,...
#> $ group <fct> A, B, B, A, A, B, B, A, A, B, B, A, B, B, A, B, A, B, A,...
# tidy data
df <- df %>%
group_by(id) %>%
mutate(from = (row_number() - 1) / n(),
to = row_number() / n()) %>%
ungroup() %>%
rowwise() %>%
mutate(list = seq(from + 1/60, to, 1/60) %>% list()) %>%
unnest()
# plot
df %>%
ggplot(aes(x = list, fill = group)) +
geom_histogram(binwidth = 1/60) +
ggthemes::theme_hc()

Created on 2018-09-16 by the [reprex package](http://reprex.tidyverse.org) (v0.2.0).
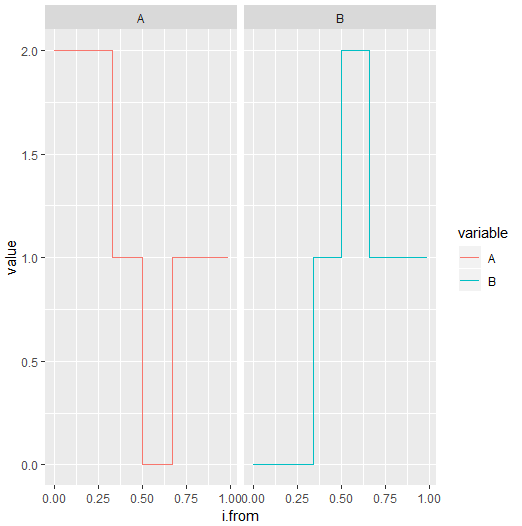
Моя попытка ответа.. хотя это, вероятно, не самый хороший / самый быстрый / самый эффективный способ, я думаю, что он может помочь вам в ваших усилиях.
library(data.table)
# compute "intervals" for each person [start, end]
df <- cj_df_raw %>%
group_by(id) %>%
mutate(pos = row_number(),
len = length(id),
from = (pos - 1) / len,
to = pos / len,
value = 1)
dt <- as.data.table(df)
setkey(dt, from, to)
#create intervals
dt.interval <- data.table(from = seq( from = 0, by = 0.01, length.out = 100),
to = seq( from = 0.01, by = 0.01, length.out = 100))
#perform overlap join on intervals
dt2 <- foverlaps( dt.interval, dt, type = "within", nomatch = NA)[, sum(value), by = c("i.from", "group")]
#some melting ans casting to fill in '0' on empty intervals
dt3 <- melt( dcast(dt2, ... ~ group, fill = 0), id.vars = 1 )
#plot
ggplot( dt3 ) +
geom_step( aes( x = i.from, y = value, color = variable ) ) +
facet_grid( .~variable )