Определяемая пользователем функция связи для glmer для моделирования выживания с известной судьбой
Распространенной ситуацией в экологии является модель выживания с бинарным исходом (0= умереть, 1= выжить), где индивидуумы (в данном примере думают об отдельных попытках гнездования птицами) различаются по количеству дней, в течение которых они подвергаются риску смертности. Чтобы учесть это, мы используем модифицированную логистическую регрессию, которая включает количество дней воздействия в функцию ссылки.
Как описано Shaffer (2004):
"Суточная выживаемость моделируется в терминах х путем выбора подходящей функции предиктора, которая в нашем случае должна давать значения от нуля до единицы. Как это делается в логистической регрессии, мы используем S-образную логистическую функцию:
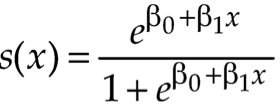
Системная составляющая нашей обобщенной линейной модели равна [s(x)]t. Далее рассмотрим функцию:
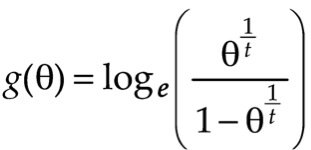
Вышеуказанная функция является монотонной и дифференцируемой относительно θ, и можно показать, что g(θ) = β0 + β1x, что удовлетворяет критериям функции связи в обобщенной линейной модели. Эти три компонента: распределение биномиального отклика, функция предиктора, заданная в выражении 1, и функция связи, заданная в выражении 2, полностью определяют нашу обобщенную линейную модель. Модель (далее "модель логистической экспозиции") аналогична модели логистической регрессии, но отличается по форме функции связи. Функция связи логистической экспозиции содержит показатель степени (1/t) в числителе и знаменателе, которого нет в функции связи логистической регрессии. Показатель степени необходим для учета того факта, что вероятность выживания в интервале зависит от длины интервала ".
Код для этой функции ссылки доступен в Интернете, а также является одной из примеров функций ссылок, описанных в R, если вы наберете "help (family)":
logexp <- function(days = 1)
{
linkfun <- function(mu) qlogis(mu^(1/days))
linkinv <- function(eta) plogis(eta)^days
mu.eta <- function(eta) days * plogis(eta)^(days-1) *
.Call("logit_mu_eta", eta, PACKAGE = "stats")
valideta <- function(eta) TRUE
link <- paste("logexp(", days, ")", sep="")
structure(list(linkfun = linkfun, linkinv = linkinv,
mu.eta = mu.eta, valideta = valideta, name = link),
class = "link-glm")
}
Это прекрасно работает в такой модели:
glm(survive ~ date, family=binomial(link=logexp(days=dat$Days)),data=dat)
Проблема, с которой я сталкиваюсь, заключается в том, что при попытке использовать эту пользовательскую функцию связи в модели GLMER с добавлением случайного эффекта (я нашел один онлайн-пример реализации этого метода здесь: http://rstudio-pubs-static.s3.amazonaws.com/4082_51aa699bd9f041c7b3f7cf7b9252f60c.html).
В нашем случае мы бы хотели включить сайт как случайный эффект. Модели формулируются так же, как и GLM из ранее:
glmer(survive ~ date + (1|site), family=binomial(link=logexp(days=dat$Days)),data=dat)
Однако теперь я получаю сообщение об ошибке:
Ошибка в famType(семейство glmFit$): неизвестная ссылка: "logexp(3)" неизвестная ссылка: "logexp(4)" неизвестная ссылка: "logexp(3)" неизвестная ссылка: "logexp(2)" неизвестная ссылка: "logexp(3) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(4) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(2) неизвестная ссылка: logexp(1) неизвестная ссылка: "logexp(4)" неизвестная ссылка: "logexp(5)" неизвестная ссылка: "logexp(4)" неизвестная ссылка: "logexp(3)" неизвестная ссылка: "logexp(4)" неизвестная ссылка: "logexp(5) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(4) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(3) неизвестная ссылка: неизвестная ссылка "logexp(3)": неизвестная ссылка "logexp(3)": неизвестная ссылка "logexp(3)": неизвестная ссылка "logexp(3)": неизвестная ссылка "logexp(3)": logexp(3)) неизвестная ссылка: logexp(2) неизвестная ссылка: logexp(1) неизвестная ссылка: logexp(3) неизвестная ссылка: logexp(1) неизвестная ссылка: logexp(1) неизвестная ссылка: "logexp(1)" неизвестная ссылка: "logexp(1)" unkn Дополнительно: Предупреждение: In if (! (lTyp <- match (семейство $ link, linkNms, nomatch = 0))) stop (gettextf ("unknow) n link:% s ",: условие имеет длину> 1, и будет использоваться только первый элемент
В сообщении об ошибке указывается неизвестная ссылка для каждой строки данных с номерами, соответствующими дням воздействия для этого посещения гнезда (или ряда данных).
пример: первый 'logexp(3)' соответствует первой строке данных, которая имеет 3 дня воздействия.
Кто-нибудь еще мог использовать эту пользовательскую функцию связи в модели GLMER? Или, если нет, у кого-нибудь есть представление о том, что является причиной ошибки?
ОБНОВИТЬБольшое спасибо Бену Болкеру за решение этой проблемы. Я обновил до 3.0.2 и последней версии lme4 и использовал функцию ссылки, связанную с Беном R ( http://rpubs.com/bbolker/4082), которая такова:
library(MASS)
logexp <- function(exposure = 1)
{
linkfun <- function(mu) qlogis(mu^(1/exposure))
## FIXME: is there some trick we can play here to allow
## evaluation in the context of the 'data' argument?
linkinv <- function(eta) plogis(eta)^exposure
mu.eta <- function(eta) exposure * plogis(eta)^(exposure-1) *
.Call(stats:::C_logit_mu_eta, eta, PACKAGE = "stats")
valideta <- function(eta) TRUE
link <- paste("logexp(", deparse(substitute(exposure)), ")",
sep="")
structure(list(linkfun = linkfun, linkinv = linkinv,
mu.eta = mu.eta, valideta = valideta,
name = link),
class = "link-glm")
}
1 ответ
Вам нужно использовать более свежую версию lme4 пакет, например, версия 1.0-4, которая только что вышла на CRAN. Более ранние версии не допускали пользовательских функций связи.
Кроме того, обратите внимание, что код, который вы разместили выше, не тот, который появляется в последних версиях ?familyгде (устарел) .Call("logit_mu_eta", eta, PACKAGE = "stats") заменяется реализацией pure-R:
logexp <- function(days = 1)
{
linkfun <- function(mu) qlogis(mu^(1/days))
linkinv <- function(eta) plogis(eta)^days
mu.eta <- function(eta) days * plogis(eta)^(days-1) * binomial()$mu_eta
valideta <- function(eta) TRUE
link <- paste0("logexp(", days, ")")
structure(list(linkfun = linkfun, linkinv = linkinv,
mu.eta = mu.eta, valideta = valideta, name = link),
class = "link-glm")
}
Ссылка, которую вы указали выше, http://rpubs.com/bbolker/4082, на самом деле имеет пример такой модели (но для нее требуется последняя версия lme4).