Выровняйте участки в ggplot
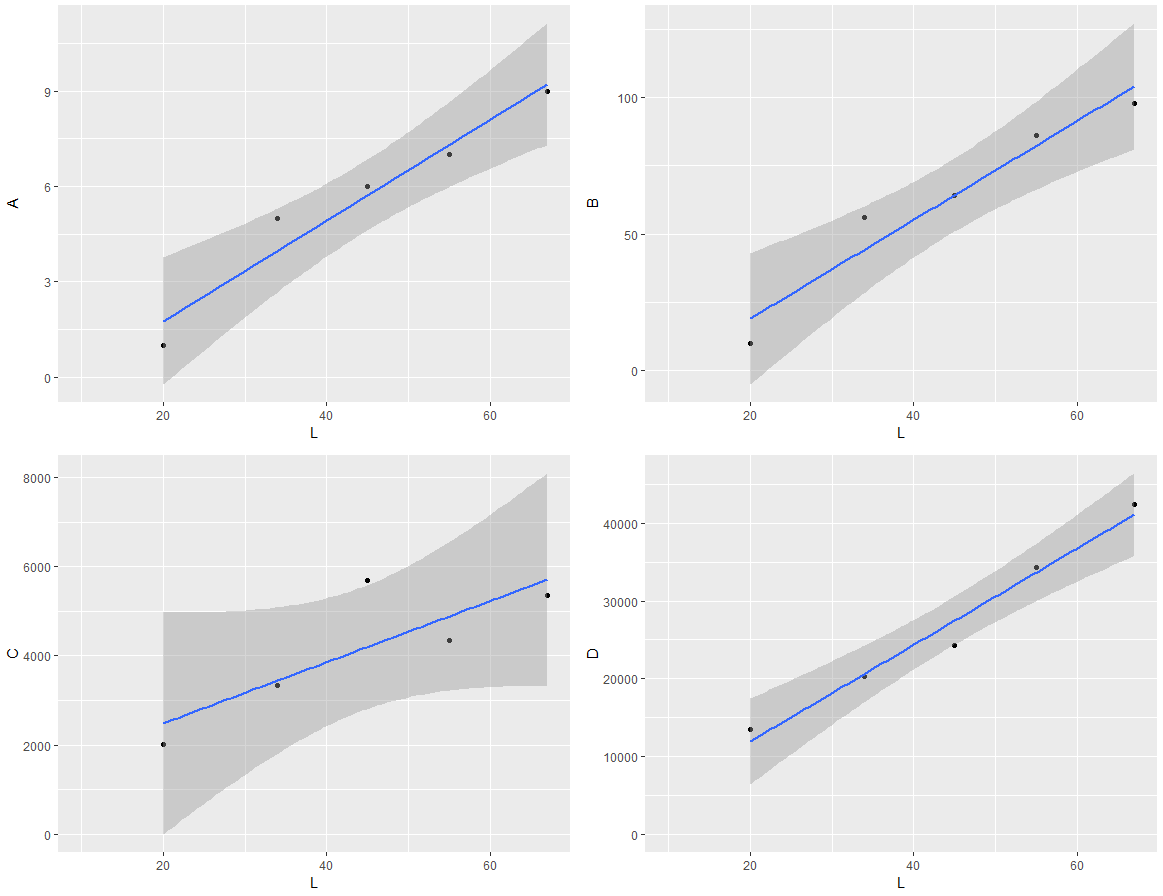
Я пытаюсь использовать grid.arrange для отображения нескольких графиков на одной странице, созданной ggplot. На графиках используются одни и те же данные x, но с разными переменными y. Графики имеют разные размеры из-за того, что у-данные имеют разные масштабы.
Я пытался использовать различные параметры темы в ggplot2, чтобы изменить размер графика и переместить метку оси y, но ни один из них не работал для выравнивания графиков. Я хочу, чтобы графики располагались в квадрате 2 x 2, чтобы каждый график был одинакового размера, а оси X совмещены.
Вот некоторые тестовые данные:
A <- c(1,5,6,7,9)
B <- c(10,56,64,86,98)
C <- c(2001,3333,5678,4345,5345)
D <- c(13446,20336,24333,34345,42345)
L <- c(20,34,45,55,67)
M <- data.frame(L, A, B, C, D)
И код, который я использую для построения:
x1 <- ggplot(M, aes(L, A,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x2 <- ggplot(M, aes(L, B,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x3 <- ggplot(M, aes(L, C,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x4 <- ggplot(M, aes(L, D,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
grid.arrange(x1,x2,x3,x4,nrow=2)
Если вы запустите этот код, вы увидите, что два нижних графика имеют меньшую площадь графика из-за большей длины единиц по оси Y.
Как мне сделать фактические окна графика одинаковыми?
5 ответов
Я бы использовал огранку для этой проблемы:
library(reshape2)
dat <- melt(M,"L") # When in doubt, melt!
ggplot(dat, aes(L,value)) +
geom_point() +
stat_smooth(method="lm") +
facet_wrap(~variable,ncol=2,scales="free")
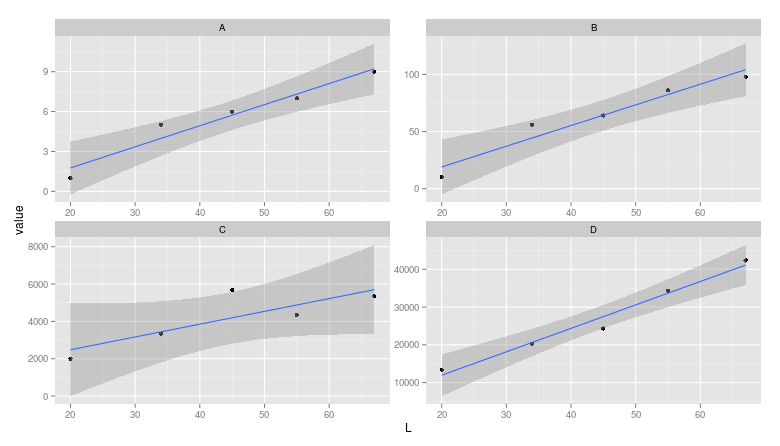
Примечание: неспециалист может не заметить, что масштабы у разных граней разные.
редактировать
Более простые решения: 1) использовать cowplot пакет (см. ответ здесь); или 2) использовать egg пакет доступен на github.
# devtools::install_github("baptiste/egg")
library(egg)
library(grid)
g = ggarrange(x1, x2, x3, x4, ncol = 2)
grid.newpage()
grid.draw(g)
оригинал
Незначительное редактирование: обновление кода.
Если вы хотите сохранить метки осей, то с некоторой путаницей и заимствованием кода отсюда это сделает свою работу.
library(ggplot2)
library(gtable)
library(grid)
library(gridExtra)
# Get the widths
gA <- ggplotGrob(x1)
gB <- ggplotGrob(x2)
gC <- ggplotGrob(x3)
gD <- ggplotGrob(x4)
maxWidth = unit.pmax(gA$widths[2:3], gB$widths[2:3],
gC$widths[2:3], gD$widths[2:3])
# Set the widths
gA$widths[2:3] <- maxWidth
gB$widths[2:3] <- maxWidth
gC$widths[2:3] <- maxWidth
gD$widths[2:3] <- maxWidth
# Arrange the four charts
grid.arrange(gA, gB, gC, gD, nrow=2)
АЛЬТЕРНАТИВНЫЕ РЕШЕНИЯ: Есть rbind а также cbind функции в gtable пакет для объединения гробов в один гроб. Для графиков здесь ширина должна быть установлена с помощью size = "max", но версия CRAN gtable выдает ошибку.
Одним из вариантов является изучение grid.arrange сюжет, а затем использовать size = "first" или size = "last"`опции:
# Get the ggplot grobs
gA <- ggplotGrob(x1)
gB <- ggplotGrob(x2)
gC <- ggplotGrob(x3)
gD <- ggplotGrob(x4)
# Arrange the four charts
grid.arrange(gA, gB, gC, gD, nrow=2)
# Combine the plots
g = cbind(rbind(gA, gC, size = "last"), rbind(gB, gD, size = "last"), size = "first")
# draw it
grid.newpage()
grid.draw(g)
Второй вариант заключается в bindфункции от gridExtra пакет.
# Get the ggplot grobs
gA <- ggplotGrob(x1)
gB <- ggplotGrob(x2)
gC <- ggplotGrob(x3)
gD <- ggplotGrob(x4)
# Combine the plots
g = cbind.gtable(rbind.gtable(gA, gC, size = "max"), rbind.gtable(gB, gD, size = "max"), size = "max")
# Draw it
grid.newpage()
grid.draw(g)
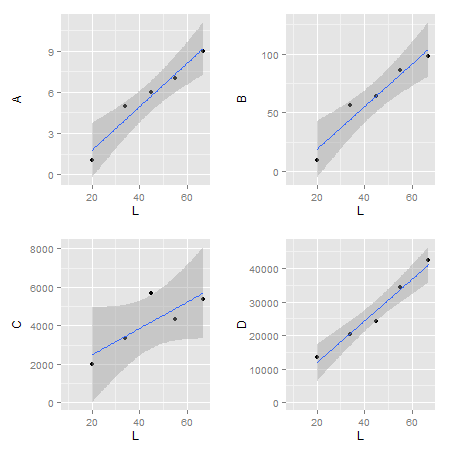
Это именно та проблема, для которой я написал пакет cowplot. Это можно сделать одной строкой в этом пакете:
require(cowplot) # loads ggplot2 as dependency
# re-create the four plots
A <- c(1,5,6,7,9)
B <- c(10,56,64,86,98)
C <- c(2001,3333,5678,4345,5345)
D <- c(13446,20336,24333,34345,42345)
L <- c(20,34,45,55,67)
M <- data.frame(L, A, B, C, D)
x1 <- ggplot(M, aes(L, A,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x2 <- ggplot(M, aes(L, B,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x3 <- ggplot(M, aes(L, C,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
x4 <- ggplot(M, aes(L, D,xmin=10,ymin=0)) + geom_point() + stat_smooth(method='lm')
# arrange into grid and align
plot_grid(x1, x2, x3, x4, align='vh')
Это результат: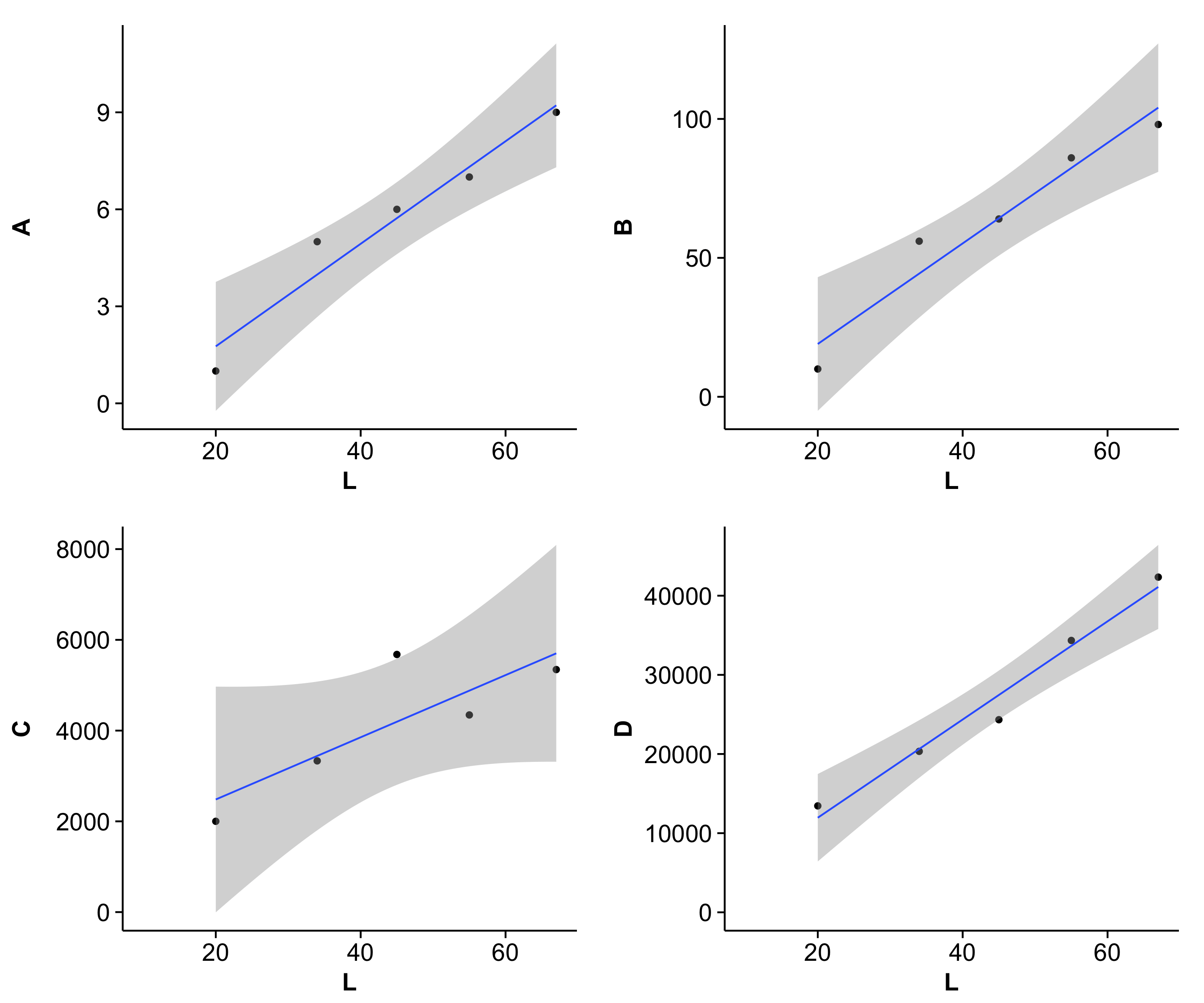 (Обратите внимание, что cowplot меняет стандартную тему ggplot2. Вы можете вернуть серую тему обратно, если действительно захотите.)
(Обратите внимание, что cowplot меняет стандартную тему ggplot2. Вы можете вернуть серую тему обратно, если действительно захотите.)
В качестве бонуса вы также можете добавить метки графиков в верхнем левом углу каждого графика:
plot_grid(x1, x2, x3, x4, align='vh', labels=c('A', 'B', 'C', 'D'))
Результат: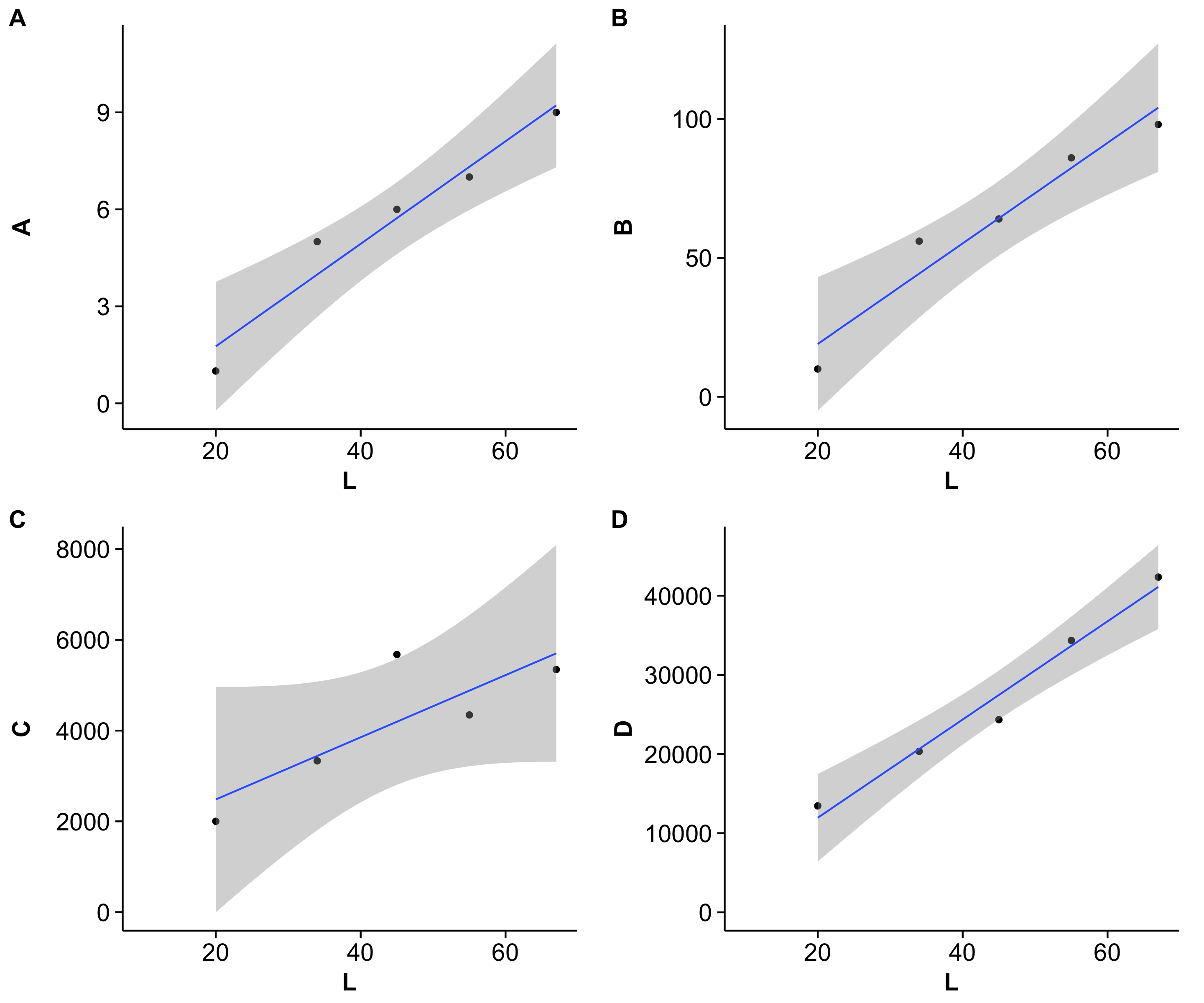
Я использую labels вариант практически на каждом графике из нескольких частей, которые я делаю.
Patchwork - это новый пакет, который позволяет легко форматировать и размещать несколько ggplots. Одним из лучших преимуществ этого является то, что он автоматически выравнивает области графика. Плюс, синтаксис действительно прост.
devtools::install_github("thomasp85/patchwork")
library(patchwork)
x1 + x2 + x3 + x4 + plot_layout(ncol = 2)
Проверьте страницу GitHub для большего количества примеров: https://github.com/thomasp85/patchwork
Если вы используете RMarkdown и вязание в PDF, у меня есть альтернативный подход. Knitr предлагает функциональность для построения подфигур при создании PDF, что позволяет вам иметь несколько рисунков на графике, каждый из которых имеет собственную подпись.
Чтобы это работало, каждый график должен отображаться отдельно. Объединив несколько функций из cowplot пакет, я сделал следующую функцию, которая выравнивает графики, сохраняя их как отдельные объекты:
plot_grid_split <- function(..., align = "hv", axis= "tblr"){
aligned_plots <- cowplot::align_plots(..., align=align, axis=axis)
plots <- lapply(1:length(aligned_plots), function(x){
cowplot::ggdraw(aligned_plots[[x]])
})
invisible(capture.output(plots))
}
Вот пример, сравнивающий макет обычно с использованием функции:
---
output: pdf_document
header-includes:
- \usepackage{subfig}
---
```{r}
plot_grid_split <- function(..., align = "hv", axis= "tblr"){
aligned_plots <- cowplot::align_plots(..., align=align, axis=axis)
plots <- lapply(1:length(aligned_plots), function(x){
cowplot::ggdraw(aligned_plots[[x]])
})
invisible(capture.output(plots))
}
```
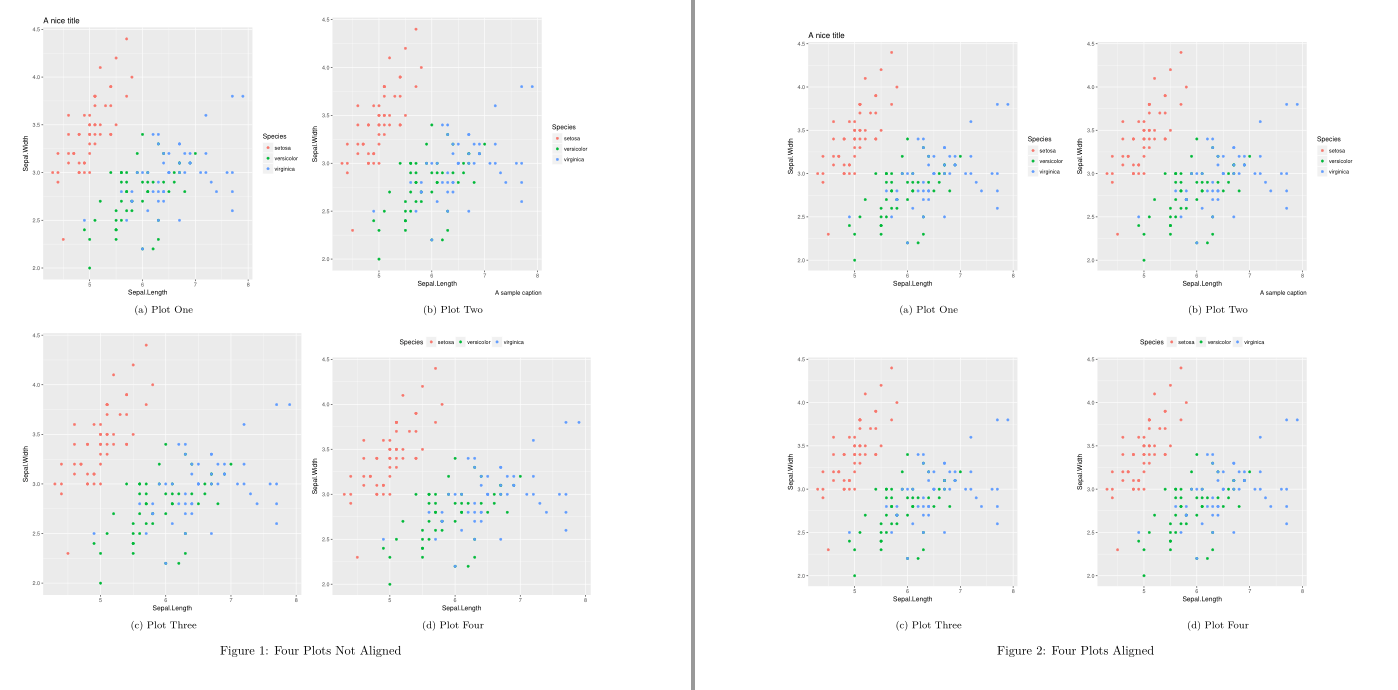
```{r fig-sub, fig.cap='Four Plots Not Aligned', fig.subcap=c('Plot One', 'Plot Two', 'Plot Three', 'Plot Four'), out.width='.49\\linewidth', fig.asp=1, fig.ncol = 2}
library(ggplot2)
plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width, colour = Species)) +
geom_point()
plot + labs(title = "A nice title")
plot + labs(caption = "A sample caption")
plot + theme(legend.position = "none")
plot + theme(legend.position = "top")
```
```{r fig-sub-2, fig.cap='Four Plots Aligned', fig.subcap=c('Plot One', 'Plot Two', 'Plot Three', 'Plot Four'), out.width='.49\\linewidth', fig.asp=1, fig.ncol = 2}
x1 <- plot + labs(title = "A nice title")
x2 <- plot + labs(caption = "A sample caption")
x3 <- plot + theme(legend.position = "none")
x4 <- plot + theme(legend.position = "top")
plot_grid_split(x1, x2, x3, x4)
```
- Вы можете узнать больше о подфигурах в R в этом посте.
- Кроме того, вы можете проверить параметры knitr, чтобы узнать больше о параметрах чанков для подфигур: https://yihui.name/knitr/options/