Использование пуассоновской структуры ошибок в нелинейном подборе
Я установил трилинейную модель
library(nlstools)
library(nlsMicrobio)
library(investr) # for plotFit function
trilinear
LOG10N ~ LOG10N0 - (t >= Sl) * (t <= (Sl + (LOG10N0 - LOG10Nres) *
log(10)/kmax)) * kmax * (t - Sl)/log(10) + (t >= Sl) * (t >
(Sl + (LOG10N0 - LOG10Nres) * log(10)/kmax)) * (LOG10Nres -
LOG10N0)
к данным бактериальной выживаемости
data(survivalcurve1)
survivalcurve1
t LOG10N
1 0.00 7.56
2 0.33 7.41
3 1.00 7.26
4 2.00 7.30
5 3.00 7.26
6 4.00 7.15
7 5.00 7.30
8 6.00 6.48
9 7.00 6.15
10 8.00 5.30
11 9.00 4.78
12 10.00 5.11
13 11.00 2.30
14 13.00 3.15
15 14.00 2.00
16 16.00 1.00
17 18.00 1.00
18 20.00 1.00
19 23.00 1.00
используя OLS в сочетании с nls:
nls = nls(trilinear, survivalcurve1,
list(Sl = 5, kmax = 1.5, LOG10N0 = 7, LOG10Nres = 1))
overview(nls)
Parameters:
Estimate Std. Error t value Pr(>|t|)
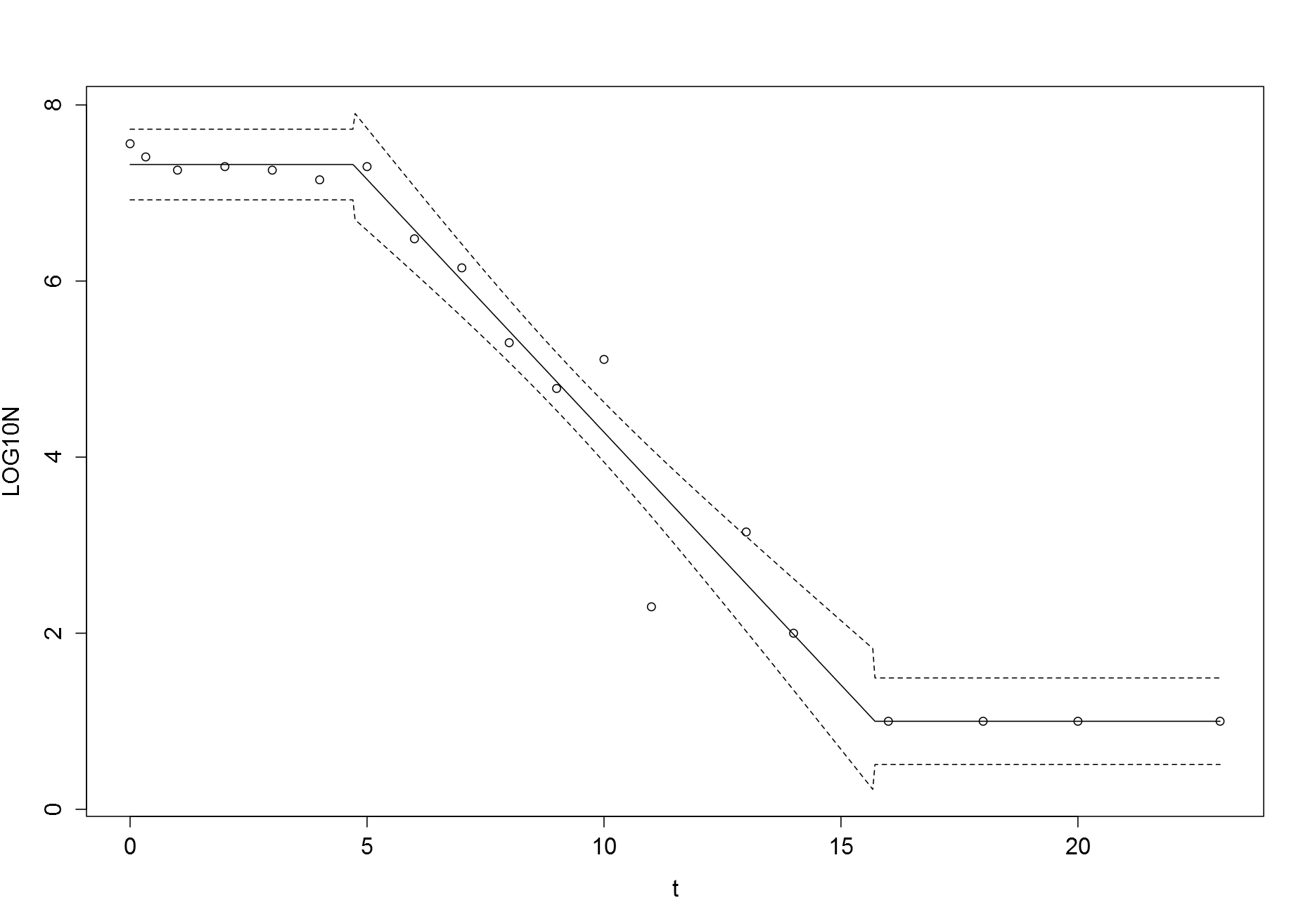
Sl 4.7064 0.5946 7.915 9.82e-07 ***
kmax 1.3223 0.1222 10.818 1.76e-08 ***
LOG10N0 7.3233 0.1884 38.875 < 2e-16 ***
LOG10Nres 1.0000 0.2307 4.334 0.00059 ***
t-based confidence interval:
2.5% 97.5%
Sl 3.4389618 5.973874
kmax 1.0617863 1.582868
LOG10N0 6.9218035 7.724863
LOG10Nres 0.5082284 1.491772
plotFit(nls, interval="confidence")
Мне было интересно, если бы я мог также соответствовать этой модели, используя максимальную вероятность для исходных (не логарифмированных) ячеек NRS (который будет в этом случае survivalcurve1$N = (10^survivalcurve1$LOG10N)), принимая во внимание, что структура ошибки будет приблизительно Пуассона? Может быть, это может быть сделано с помощью bbmle"s mle2и если так, какой будет правильный синтаксис?
РЕДАКТИРОВАТЬ: я пытался с
survivalcurve1$N = as.integer(10^survivalcurve1$LOG10N)
trilinearN=formula(N ~ dpois( 10^(LOG10N0 - (t >= Sl) * (t <= (Sl + (LOG10N0 - LOG10Nres) *
log(10)/kmax)) * kmax * (t - Sl)/log(10) + (t >= Sl) * (t > (Sl + (LOG10N0 - LOG10Nres) * log(10)/kmax)) * (LOG10Nres - LOG10N0))))
m1 = mle2(trilinearN, start=list(Sl = 5, kmax = 1.5, LOG10N0 = 7, LOG10Nres = 1), data=survivalcurve1)
а также
coef(summary(m1))
дает мне
Estimate Std. Error z value Pr(z)
Sl 4.902048 1.669354e-04 2.936495e+04 0
kmax 1.475309 3.210865e-04 4.594739e+03 0
LOG10N0 7.344014 3.785883e-05 1.939842e+05 0
LOG10Nres -1.830498 1.343019e-10 -1.362972e+10 0
Не удалось заставить работать прогнозы:
df=data.frame(t=seq(0,max(survivalcurve1$t),length=100))
df$pred=predict(m1,newdata=df)
with(df,lines(t,pred,col=2))
как это дало мне ошибку
Error : object of type 'symbol' is not subsettable
Error in gfun(object, newdata = newdata, location = location, op = "predict") :
can only use predict() if formula specified
Какие-нибудь мысли? Кроме того, как бы я разобрать, если Пуассон mle2 подходит лучше, чем nls один? (Поскольку AIC нельзя сравнивать из-за разницы в масштабе)
PS The geeraerd модель тоже подойдет, если будет проще:
geeraerd
LOG10N ~ LOG10Nres + log10((10^(LOG10N0 - LOG10Nres) - 1) * exp(kmax *
Sl)/(exp(kmax * t) + (exp(kmax * Sl) - 1)) + 1)