Веса в glmmPQL говорят, что переменные длины: R
Люди касаются поверхностей и собирают микробы на пальцах. Я измеряю микробы на их пальцах после X числа поверхностных контактов (NumberContacts) (фиксированный эффект). Это зависит от участника (случайный эффект), а также от того, носят ли они перчатки или нет (перчатки) (фиксированный эффект). Вот необработанные данные: 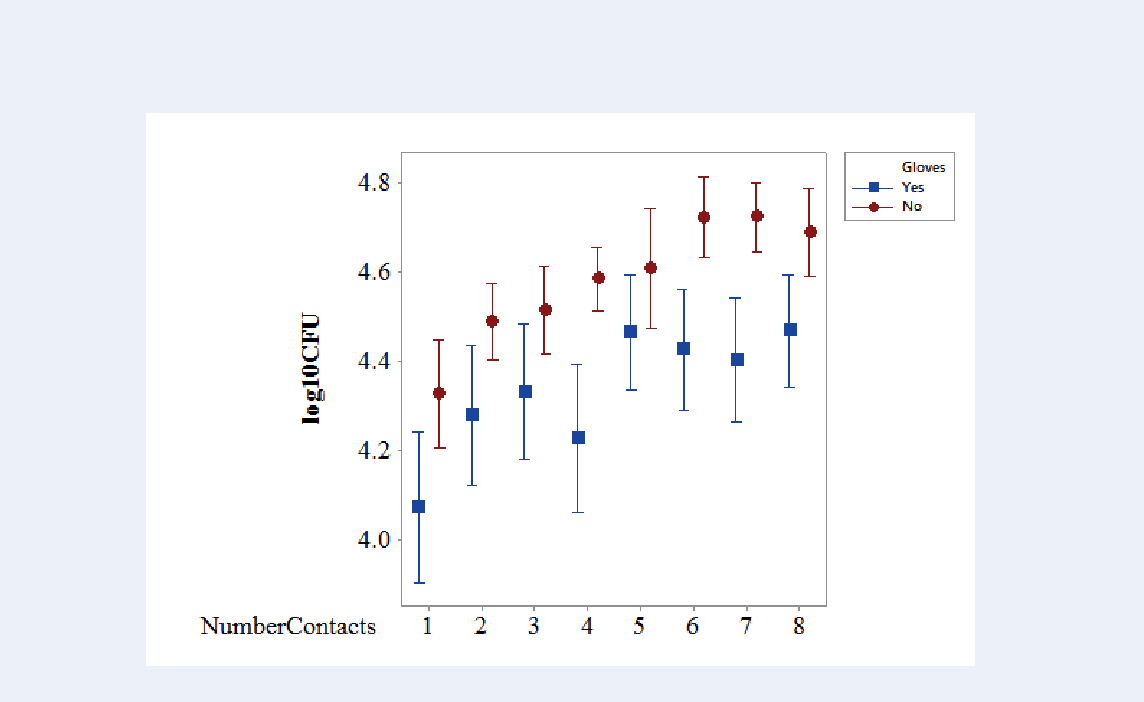
Я пытаюсь запустить обобщенную линейную смешанную модель с PQL, используя пакет MASS в R, потому что мои данные ненормальны. Я преобразовал их, используя преобразование Бокса-Кокса с lambda=0,779, но думаю, что это похоже на нормальное логарифмическое распределение, поэтому использовал функцию log-link.
K<-data.fame(germs=rlnorm(560,4,5),
Participant=rep(1:35,each=16),
NumberContacts=rep(1:8,70),
Gloves=rep(c("G","U"),70))
PQL <- glmmPQL(germs^0.779 ~ Gloves+NumberContacts, ~1 | Participant, family = gaussian(link = "log"),
data = na.omit(Ksub),
verbose = F)
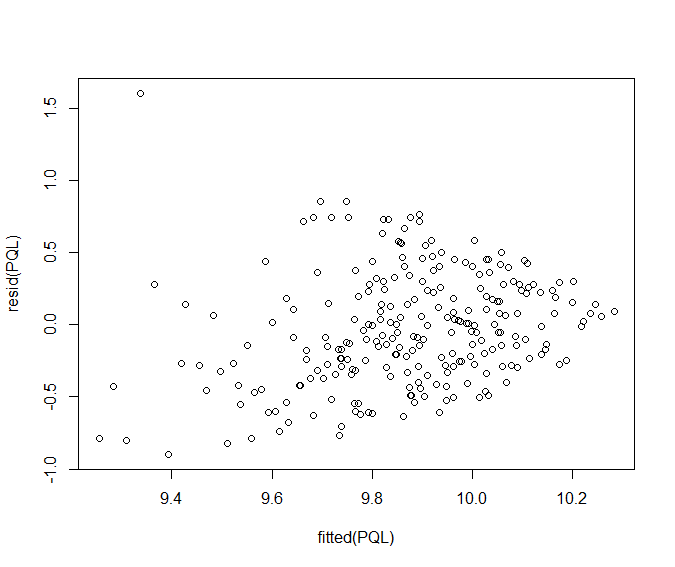
Но я также думаю, что мои отклонения не одинаковы по остаточным значениям (см. Число остатков в сравнении с установленным рисунком). Сказав это, тест Левена отвергает гетероскедастичность в моих данных между группами (р = 0,5). Но посмотрите на остатки против установлены. Я хотел бы объяснить это, используя взвешенные отклонения.
PQL <- glmmPQL(germs^0.779 ~ Gloves+poly(NumberContacts,2), ~1 | Participant, family = gaussian(link = "log"),
weights =varIdent(form = ~1 | NumberContacts),
data = na.omit(Ksub),
verbose = F)
Я добавил weights=varIdent(form = ~1 | NumberContacts) по этой ссылке varIdent, но получаю ошибку:
Ошибка в model.frame.default(data = na.omit(Ksub), weights = varIdent(form = ~1 переменной длины отличаются (найдено для '(weights)')
Почему это? Также не стесняйтесь комментировать что-либо еще, что выглядит странно (например, преобразование данных!).