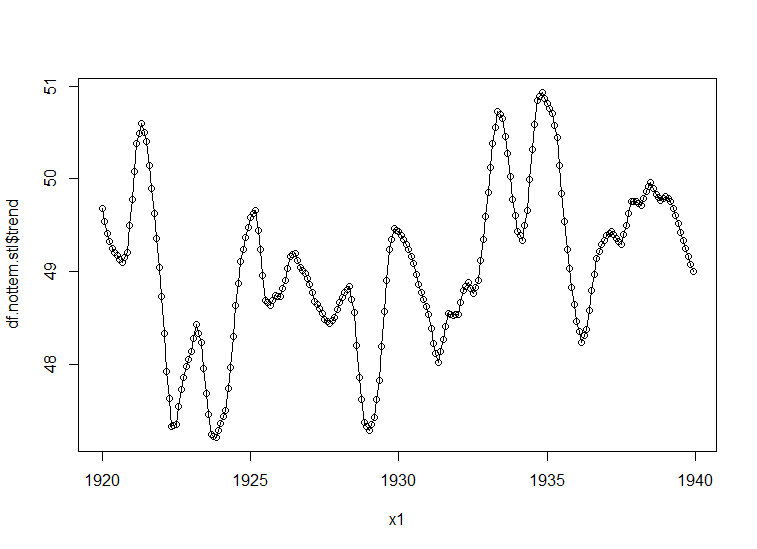Разложение LOESS (STL) с использованием Ggvis
Я хочу иметь возможность построить три различных элемента разложения сезонных трендов, используя Лёсс (STL) с Ggvis.
Тем не менее, я получаю эту ошибку:
Error: data_frames can only contain 1d atomic vectors and lists
Я использую набор данных nottem.
# The Seasonal Trend Decomposition using Loess (STL) with Ggvis
# Load nottem data set
library(datasets)
nottem <- nottem
# Decompose using stl()
nottem.stl = stl(nottem, s.window="periodic")
# Plot decomposition
plot(nottem.stl)
Теперь это информация, которая меня интересует. Чтобы превратить это в график, с которым я могу поиграть, я преобразую его в фрейм данных с помощью пакета xts. Все идет нормально.
# Transform nottem.stl to a data.frame
library(xts)
df.nottem.stl <- as.data.frame(as.xts(nottem.stl$time.series))
# Add date to data.frame
df.nottem.stl$date <- data.frame(time = seq(as.Date("1920-01-01"), by = ("months"), length =240))
# Glimpse data
glimpse(df.nottem.stl)
# Plot simple line of trend
plot(df.nottem.stl$date, df.nottem.stl$trend, type = "o")
Это в значительной степени сюжет, который я хочу. Тем не менее, я хочу иметь возможность использовать его с Shiny, и поэтому Ggvis предпочтительнее.
# Plot ggvis
df.nottem.stl%>%
ggvis(~date, ~trend)%>%
layer_lines()
Вот где я получаю свою ошибку.
Любые намеки на то, что может пойти не так?
1 ответ
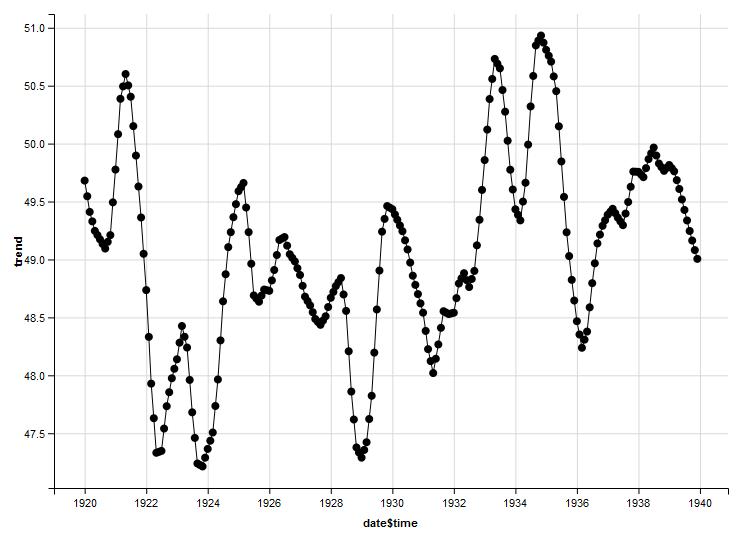
Прежде всего ваш df.nottem.stl data.frame содержит Date data.frame, так что вы должны использовать date$time колонка. Затем с помощью layer_paths функция вместо layer_lines заставит это работать. Я всегда нахожу layer_paths работает лучше чем layer_lines:
Так что это будет работать:
library(ggvis)
df.nottem.stl%>%
ggvis(~date$time, ~trend)%>%
#for points
layer_points() %>%
#for lines
layer_paths()
Выход: