Библиотека "TableOne" несколько сравнений. Рассчитать построчно p-значения
Я получил комментарий от рецензента, который хотел иметь все p-значения для каждой строки уровней определенных переменных в таблице демографических характеристик (таблица 1). Хотя эта просьба кажется мне довольно странной (и неточной), я хотел бы согласиться с его предложением.
library(tableone)
## Load data
library(survival); data(pbc)
# drop ID from variable list
vars <- names(pbc)[-1]
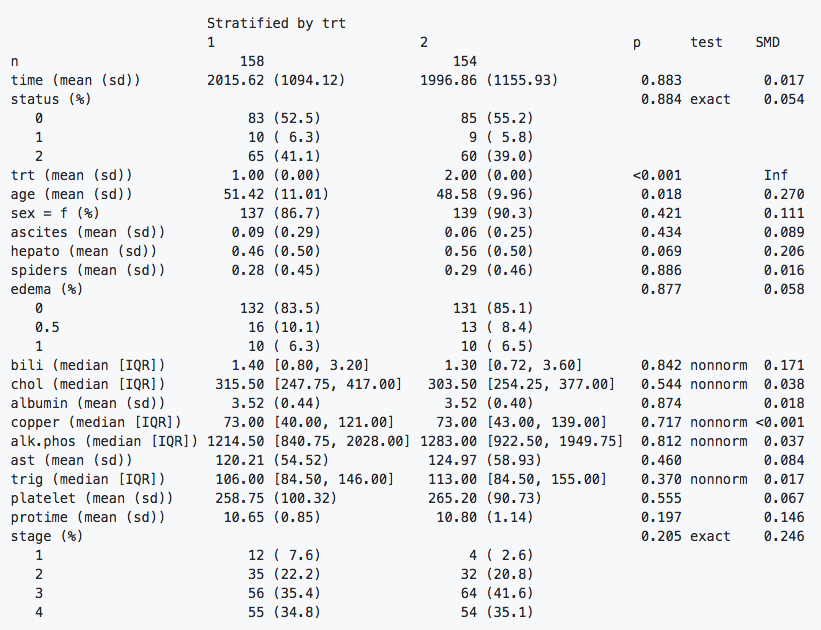
## Create Table 1 stratified by trt (can add more stratifying variables)
tableOne <- CreateTableOne(vars = vars, strata = c("trt"), data = pbc, factorVars = c("status","edema","stage"))
print(tableOne, nonnormal = c("bili","chol","copper","alk.phos","trig"), exact = c("status","stage"), smd = TRUE)
Мне нужно иметь p-значения для каждого уровня переменных status, edema а также stageс коррекцией Бонферрони. Я прошел документацию без успеха. Кроме того, правильно ли использовать хи-квадрат для сравнения размеров выборки по строкам?
ОБНОВИТЬ:
Я не уверен, что мой подход правильный, но я хотел бы поделиться им с вами. Я сгенерировал для переменной status фиктивная переменная для каждой страты, чем я рассчитал chisq,
library(tableone)
## Load data
library(survival); data(pbc)
d <- pbc[,c("status", "trt")]
# Convert dummy variables
d$status.0 <- ifelse(d$status==0, 1,0)
d$status.1 <- ifelse(d$status==1, 1,0)
d$status.2 <- ifelse(d$status==2, 1,0)
t <- rbind(
chisq.test(d$status.0, d$trt),
# p-value = 0.7202
chisq.test(d$status.1, d$trt),
# p-value = 1
chisq.test(d$status.2, d$trt)
#p-value = 0.7818
)
t
BONFERRONI ADJ ДЛЯ НЕСКОЛЬКИХ СРАВНЕНИЙ:
p <- t[,"p.value"]
p.adjust(p, method = "bonferroni")
0 ответов
Этот вопрос был опубликован некоторое время назад, поэтому я полагаю, вы уже ответили рецензенту.
Я действительно не понимаю, зачем вычислять скорректированные значения p только для трех переменных. Фактически, настройка значений p зависит от количества сделанных сравнений. Если вы используетеp.adjust() с вектором из 3 значений p результаты на самом деле не будут "скорректированы" на количество сделанных сравнений (вы действительно сделали более полутора дюжин!)
Я показываю, как извлечь все p-значения, чтобы вы могли вычислить скорректированные. Чтобы извлечь pValues из пакетаtableOne есть способ вызова атрибутов объекта (объяснено первым) и два быстрых и грязных способа (в нижней части).
Чтобы извлечь их, я сначала скопирую ваш код для создания tableOne:
library(tableone)
## Load data
library(survival); data(pbc)
# drop ID from variable list
vars <- names(pbc)[-1]
## Create Table 1 stratified by trt (can add more stratifying variables)
tableOne <- CreateTableOne(vars = vars, strata = c("trt"), data = pbc, factorVars = c("status","edema","stage"))
Вы можете увидеть, что есть у вашего объекта tableOne, через attributes()
attributes(tableOne)
Вы можете увидеть tableOne обычно имеет таблицу для непрерывных и категориальных переменных. Ты можешь использоватьattributes() в них тоже
attributes(tableOne$CatTable)
# you can notice $pValues
Теперь вы знаете, где находятся значения pValues, и можете извлечь их с помощью attr()
attr(tableOne$CatTable, "pValues")
Нечто подобное с числовыми переменными:
attributes(tableOne$ContTable)
# $pValues are there
attr(tableOne$ContTable, "pValues")
У вас есть pValues для нормальных и ненормальных переменных. Как вы установили ранее, вы можете извлечь оба
mypCont <- attr(tableOne$ContTable, "pValues") # put them in an object
nonnormal = c("bili","chol","copper","alk.phos","trig") # copied from your code
mypCont[rownames(mypCont) %in% c(nonnormal), "pNonNormal"] # extract NonNormal
"%!in%" <- Negate("%in%")
mypCont[rownames(mypCont) %!in% c(nonnormal), "pNonNormal"] # extract Normal
После всего сказанного и извлеченных pValues я думаю, что есть два более удобных быстрых и грязных способа сделать то же самое:
Быстрый и грязный способ A: использованиеdput()с вашим напечатанным tableOne. Затем найдите в консоли, где находятся pValues, и скопируйте и вставьте их в скрипт, чтобы сохранить их в объекте.
Быстрый и грязный способ B: Если вы посмотрите в tableOne vignette, там есть раздел "Экспорт", вы можете использоватьprint(tableOne, quote = TRUE)а затем просто скопируйте и вставьте в электронную таблицу (например, LibreOffice, Excel...). Затем я бы выбрал столбец с pValue, транспонировал его и вернул обратно в R, чтобы вычислить скорректированные значения p с помощьюp.adjust() и скопируйте их обратно в электронную таблицу для отправки в журнал