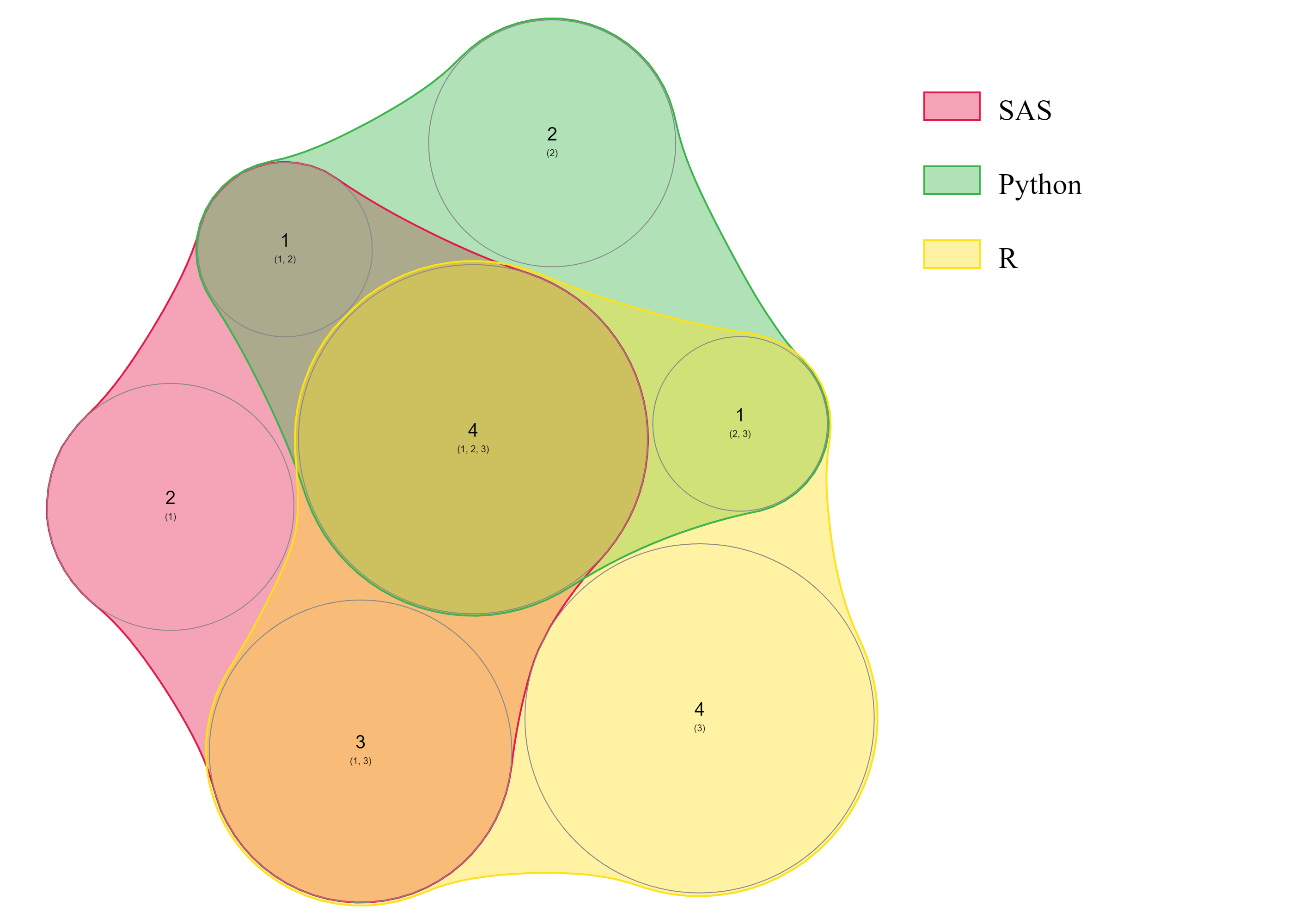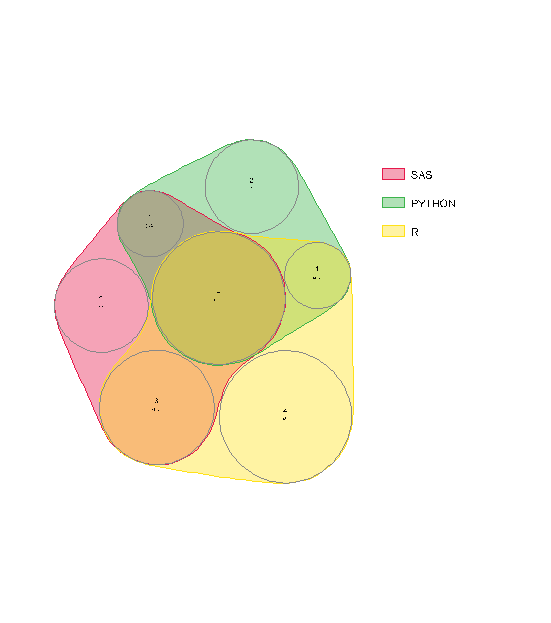Преобразование данных для создания обобщенных, квази-пропорциональных диаграмм Венна с использованием пакета 'nVennR'
У меня есть приведенный ниже набор данных, и я хотел бы, чтобы вы помогли его преобразовать, чтобы иметь возможность построить диаграмму Венна с использованием пакета 'nVennR' Переса-Силвы и соавт. 2018
Вот набор данных:
dput(data)
structure(list(Employee = c("A001", "A002", "A003", "A004", "A005",
"A006", "A007", "A008", "A009", "A010", "A011", "A012", "A013",
"A014", "A015", "A016", "A017", "A018"), SAS = c("Y", "N", "Y",
"Y", "Y", "Y", "N", "Y", "N", "N", "Y", "Y", "Y", "Y", "N", "N",
"N", "N"), Python = c("Y", "Y", "Y", "Y", "N", "N", "N", "N",
"N", "N", "Y", "Y", "N", "N", "N", "N", "Y", "Y"), R = c("Y",
"Y", "N", "Y", "N", "Y", "N", "N", "Y", "Y", "Y", "Y", "Y", "Y",
"Y", "Y", "N", "N")), .Names = c("Employee", "SAS", "Python",
"R"), row.names = c(NA, -18L), class = c("tbl_df", "tbl", "data.frame"
))
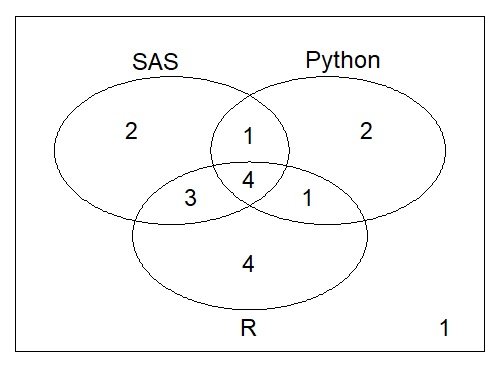
Ниже приведен пример диаграммы Венна, которую я хотел бы получить:
Обновить:
После установки обновленной версии nVennR а также rsvg, когда я запускаю пример кода отсюда, я получаю ошибку и диаграмму ниже:
Warning message:
In checkValidSVG(doc, warn = warn) :
This picture was not generated by the 'grConvert' package, errors may result
Ниже информация о моей сессии:
sessionInfo()
R version 3.4.2 (2017-09-28)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows >= 8 x64 (build 9200)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252
[2] LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252
[4] LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods
[7] base
other attached packages:
[1] nVennR_0.2.0
loaded via a namespace (and not attached):
[1] Rcpp_0.12.16 lattice_0.20-35 XML_3.98-1.10
[4] png_0.1-7 rsvg_1.1 grid_3.4.2
[7] plyr_1.8.4 gtable_0.2.0 scales_0.5.0.9000
[10] ggplot2_2.2.1.9000 pillar_1.2.1 rlang_0.2.0.9001
[13] grImport2_0.1-2 lazyeval_0.2.1 Matrix_1.2-12
[16] tools_3.4.2 munsell_0.4.3 jpeg_0.1-8
[19] compiler_3.4.2 base64enc_0.1-3 colorspace_1.3-2
[22] tibble_1.4.2
Буду признателен за любые идеи по решению этой проблемы.
4 ответа
Просто небольшое замечание, чтобы вы знали, что новая версия nVennR готова. Управление входом и выходом теперь отличается, и toVenn устарела, подлежит замене plotVenn, Здесь есть виньетка с несколькими примерами, один из которых использует данные этого вопроса.
Вот один из способов использования limma пакет в Bioconductor с вашими данными, загруженными из dput как переменная z:
source("http://www.bioconductor.org/biocLite.R")
biocLite("limma")
library(limma)
Измените все Y на TRUE и все N на FALSE:
z2 <- data.frame(lapply(z, function(x) { gsub("Y", "TRUE", x) }))
z3 <- data.frame(lapply(z2, function(x) { gsub("N", "FALSE", x) }),stringsAsFactors=FALSE)
Убедитесь, что все они имеют логический тип:
z3$SAS <- as.logical(z3$SAS)
z3$Python <- as.logical(z3$Python)
z3$R <- as.logical(z3$R)
Теперь подсчитайте все суммы по каждой области Венны, используя vennCounts:
> ( venn.totals <- vennCounts(z3[,-1]) )
SAS Python R Counts
1 0 0 0 1
2 0 0 1 4
3 0 1 0 2
4 0 1 1 1
5 1 0 0 2
6 1 0 1 3
7 1 1 0 1
8 1 1 1 4
attr(,"class")
[1] "VennCounts"
Создание диаграммы - это еще один шаг:
vennDiagram(venn.totals)
Приятно иметь обратную связь так быстро. Возможно, мы должны были заявить в документе, что эта версия nVennR является предварительной. Некоторые исследователи просили о быстром способе запуска nVenn, поэтому я просто упаковал код C++ в пару функций R. Как видите, результат показан на viewer окно вместо plot окно. Я учусь, как я иду. Поскольку я вижу некоторый интерес к этому пакету, я составляю список функций, которые необходимо добавить в следующую версию. Лучшие варианты ввода, безусловно, в этом списке. Кроме того, больше контроля над выходом (кстати, если цвета мешают, вы можете просто установить opacity до 0).
Что касается вопроса, @mysteRious прав, вы отправляете списки в функцию. Быстрый способ сделать это будет
sas <- subset(data, SAS == "Y")$Employee
python <- subset(data, Python == "Y")$Employee
rr <- subset(data, R == "Y")$Employee
mySVG <- toVenn(sas, python, rr)
showSVG(mySVG = mySVG, opacity = 0.1)
В следующей версии будет метод для ввода имен отдельно (извините за это)
Что касается меток, краткий ответ заключается в том, что вы можете редактировать их самостоятельно с помощью SVG-редактора, такого как Inkscape. Если он установлен, вы можете открыть рисунок в редакторе, запустив showSVG(mySVG = mySVG, opacity = 0.1, systemShow=T), Вы также можете сохранить рисунок, предоставив выходной файл (outFile) или просто откройте временный файл, который генерируется.
Несколько более длинный ответ заключается в том, что name1, name2,... можно заменить именами списков. К сожалению, из-за моих ограничений в R, я не понял, что это может быть не так просто. Было бы проще загрузить каждую переменную в виде таблицы и установить colNames. Например,
sas <- as.table(subset(data, SAS == "Y")$Employee)
names(sas) <- 'SAS'
Этот ярлык будет использоваться в легенде. Что касается небольших ярлыков, в настоящее время у пользователя нет возможности их изменить. Они предназначены для того, чтобы помочь определить местоположение определенных регионов, и когда эти регионы малы, кажется невозможным использовать более длинные метки. Мой совет - всегда использовать внешний редактор для их изменения. В будущей версии будет, по крайней мере, возможность удалять эти ярлыки, как в веб-версии.