Перебирайте файлы netcdf и запускайте вычисления - Python или R
Это мой первый раз, когда я использую netCDF, и я стараюсь работать с ним.
У меня есть несколько файлов netcdf версии 3 (ежедневные средние значения NOAA NARR air.2m за целый год). Каждый файл охватывает год в период с 1979 по 2012 год. Это сетки размером 349 x 277 с разрешением приблизительно 32 км. Данные были загружены отсюда.
Измерение - это время (часы с 1 января 1800 года), а моя переменная интереса - воздух. Мне нужно рассчитать накопленные дни с температурой < 0. Например
Day 1 = +4 degrees, accumulated days = 0
Day 2 = -1 degrees, accumulated days = 1
Day 3 = -2 degrees, accumulated days = 2
Day 4 = -4 degrees, accumulated days = 3
Day 5 = +2 degrees, accumulated days = 0
Day 6 = -3 degrees, accumulated days = 1
Мне нужно хранить эти данные в новом файле netcdf. Я знаком с Python и немного с R. Что является лучшим способом циклически проходить через каждый день, проверять значение предыдущих дней и, основываясь на этом, выводить значение в новый файл netcdf с точно таким же измерением и переменной... или, возможно, просто добавьте другую переменную в исходный файл netcdf с выводом, который я ищу.
Лучше ли оставить все файлы отдельно или объединить их? Я объединил их с ncrcat, и он работал нормально, но файл 2.3 ГБ.
Спасибо за вклад.
Мой текущий прогресс в Python:
import numpy
import netCDF4
#Change my working DIR
f = netCDF4.Dataset('air7912.nc', 'r')
for a in f.variables:
print(a)
#output =
lat
long
x
y
Lambert_Conformal
time
time_bnds
air
f.variables['air'][1, 1, 1]
#Output
298.37473
Чтобы лучше понять, с какой структурой данных я работаю? Является ли ['air'] клавишей в приведенном выше примере, а [1,1,1] также являются клавишами? чтобы получить значение 298,37473. Как я могу затем пройти через [1,1,1]?
3 ответа
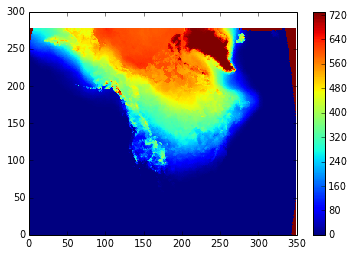
Вы можете использовать замечательную функцию MFDataset в netCDF4 для обработки группы файлов как одного агрегированного файла без необходимости использования ncrcat, Итак, ваш код будет выглядеть так:
from pylab import *
import netCDF4
f = netCDF4.MFDataset('/usgs/data2/rsignell/models/ncep/narr/air.2m.19??.nc')
# print variables
f.variables.keys()
atemp = f.variables['air']
print atemp
ntimes, ny, nx = shape(atemp)
cold_days = zeros((ny,nx),dtype=int)
for i in xrange(ntimes):
cold_days += atemp[i,:,:].data-273.15 < 0
pcolormesh(cold_days)
colorbar()
И вот один из способов записи файла (могут быть более простые способы):
# create NetCDF file
nco = netCDF4.Dataset('/usgs/data2/notebook/cold_days.nc','w',clobber=True)
nco.createDimension('x',nx)
nco.createDimension('y',ny)
cold_days_v = nco.createVariable('cold_days', 'i4', ( 'y', 'x'))
cold_days_v.units='days'
cold_days_v.long_name='total number of days below 0 degC'
cold_days_v.grid_mapping = 'Lambert_Conformal'
lono = nco.createVariable('lon','f4',('y','x'))
lato = nco.createVariable('lat','f4',('y','x'))
xo = nco.createVariable('x','f4',('x'))
yo = nco.createVariable('y','f4',('y'))
lco = nco.createVariable('Lambert_Conformal','i4')
# copy all the variable attributes from original file
for var in ['lon','lat','x','y','Lambert_Conformal']:
for att in f.variables[var].ncattrs():
setattr(nco.variables[var],att,getattr(f.variables[var],att))
# copy variable data for lon,lat,x and y
lono[:]=f.variables['lon'][:]
lato[:]=f.variables['lat'][:]
xo[:]=f.variables['x'][:]
yo[:]=f.variables['y'][:]
# write the cold_days data
cold_days_v[:,:]=cold_days
# copy Global attributes from original file
for att in f.ncattrs():
setattr(nco,att,getattr(f,att))
nco.Conventions='CF-1.6'
nco.close()
Если я попытаюсь посмотреть полученный файл в графическом интерфейсе Unidata NetCDF-Java Tools-UI, то все будет в порядке: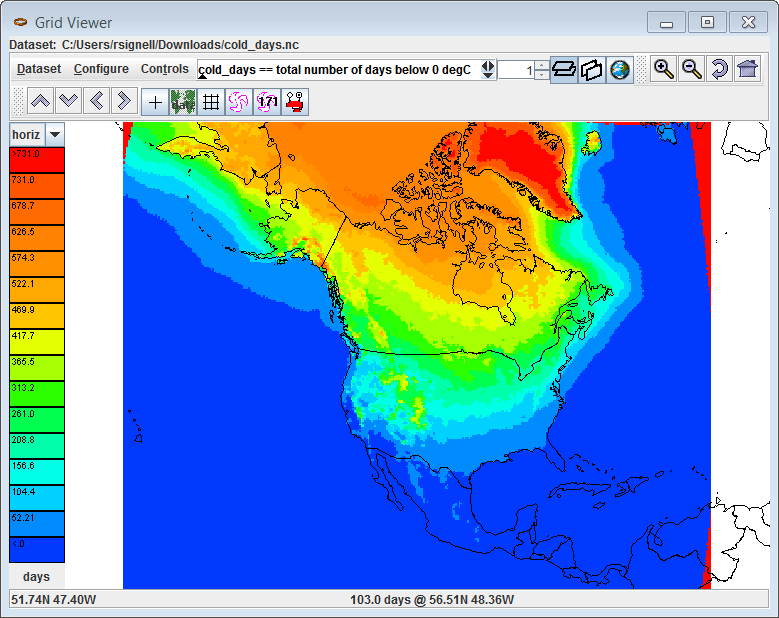 Также обратите внимание, что здесь я только что скачал два набора данных для тестирования, поэтому я использовал
Также обратите внимание, что здесь я только что скачал два набора данных для тестирования, поэтому я использовал
f = netCDF4.MFDataset('/usgs/data2/rsignell/models/ncep/narr/air.2m.19??.nc')
В качестве примера. Для всех данных, вы можете использовать
f = netCDF4.MFDataset('/usgs/data2/rsignell/models/ncep/narr/air.2m.????.nc')
или же
f = netCDF4.MFDataset('/usgs/data2/rsignell/models/ncep/narr/air.2m.*.nc')
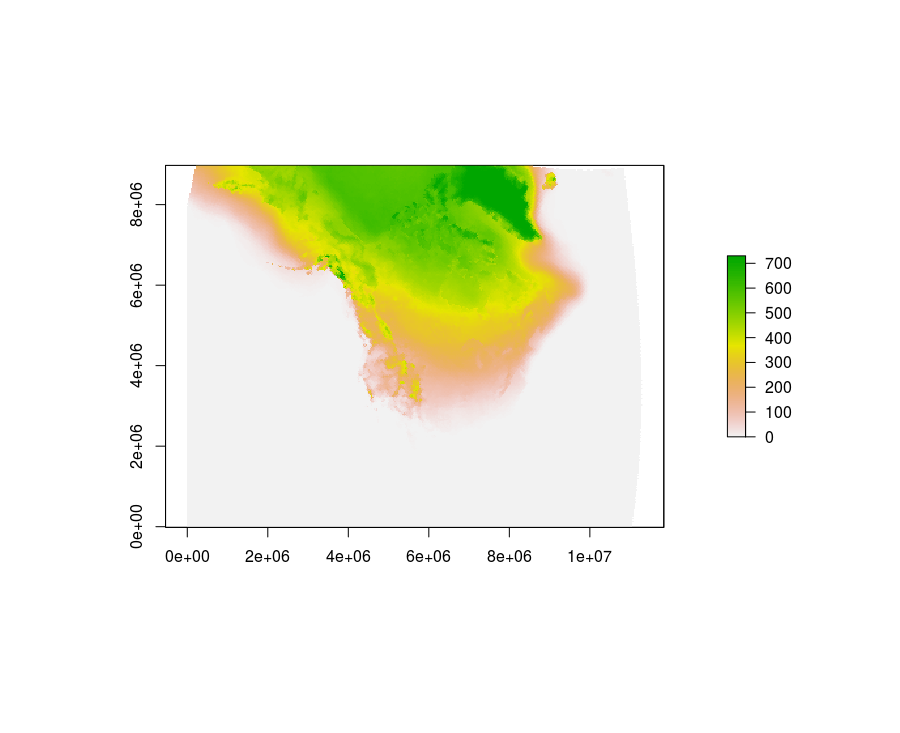
Вот R решение.
infiles <- list.files("data", pattern = "nc", full.names = TRUE, include.dirs = TRUE)
outfile <- "data/air.colddays.nc"
library(raster)
r <- raster::stack(infiles)
r <- sum((r - 273.15) < 0)
plot(r)
Я знаю, что это довольно поздно для этой темы с 2013 года, но я просто хочу отметить, что принятое решение не обеспечивает решение поставленного вопроса. Кажется, что вопрос требует продолжительности каждого непрерывного периода температур ниже нуля (обратите внимание, что счетчик сбрасывает счетчик, если температура превышает ноль), что может быть важно для климатических применений (например, для сельского хозяйства), тогда как принятое решение дает только общее количество дней в году, когда температура ниже нуля. Если это действительно то, чего хочет mkmitchell (он был принят как ответ), то это можно сделать из командной строки в cdo, не беспокоясь о входе / выходе NETCDF:
cdo timsum -lec,273.15 in.nc out.nc
так что зацикленный скрипт будет:
files=`ls *.nc` # pick up all the netcdf files in a directory
for file in $files ; do
# I use 273.15 as from the question seems T is in Kelvin
cdo timsum -lec,273.15 $file ${file%???}_numdays.nc
done
Если вам нужно общее количество за весь период, вы можете вместо этого отследить файлы _numdays, которые намного меньше:
cdo cat *_numdays.nc total.nc
cdo timsum total.nc total_below_zero.nc
Но опять же, вопрос, кажется, требует накопленных дней на событие, которое отличается, но не обеспечивается принятым ответом.