Python scikit-DBSCAN: неправильная координата или кластеризация
Я пишу DBSCAN, и я столкнулся с какой-то странной проблемой.(2 проблемы)
Вот мой код:
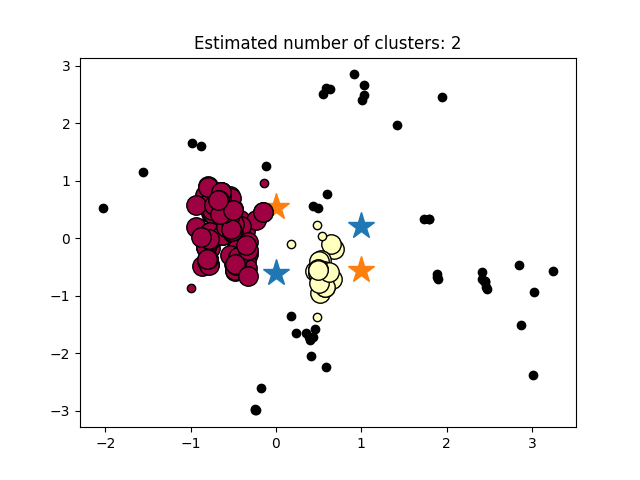
Первая часть есть проблема, если я добавлюX = StandardScaler().fit_transform(X) координата результата неверна! Но если я не добавлю этот код, это всегда будет одна кластеризация (но координата результата верна!). я пытаюсь настроить esp или min_samples, но это не изменилось.
dataSet = []
fileIn = open('data')
for line in fileIn.readlines():
lineArr = line.strip().split('\t')
dataSet.append([float(lineArr[0]), float(lineArr[1])])
numSamples = len(dataSet)
X = np.array(dataSet)
X = StandardScaler().fit_transform(X)
 (добавлять)
(добавлять) (без)
(без)
db = DBSCAN(eps=0.5, min_samples=10).fit(X)
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
print(labels)
counters = {}
for item in labels:
if item in counters:
counters[item] += 1
else:
counters[item] = 1
print ("Count of different cluster:(#r,g,b,a)")#r,g,b,a
print (counters)
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
print('Estimated number of clusters: %d' % n_clusters_)
Вторая проблема заключается в том, что я пытаюсь построить координату, которую я вычисляю, но я не знаю, почему она показывает неправильный результат!
clusters = [np.mean(X[labels == i],axis=0) for i in range(n_clusters_)]
outliers = X[labels == 0]
print(clusters)
for i in range(n_clusters_):
plt.plot(clusters[i],'*',markersize=20)
unique_labels = set(labels)
colors = [plt.cm.Spectral(each)
for each in np.linspace(0, 1, len(unique_labels))]
for k, col in zip(unique_labels, colors):
if k == -1:
# Black used for noise.
col = [0, 0, 0, 1]
class_member_mask = (labels == k)
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),markeredgecolor='k', markersize=14)
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),markeredgecolor='k', markersize=6)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
Пожалуйста, помогите мне поблагодарить вас!
1 ответ
Очевидно, вам нужно выбрать эпсилон, соответствующий вашей системе координат. Если вы масштабируете данные, эпсилон больше не будет прежним. Хак, который вы найдете более простым (но это не "правильное" решение), состоит в простом вычислении среднего значения с использованием немасштабированных данных. Но средства кластеров DBSCAN в любом случае ненадежны.
Судя по вашим осям, вам, вероятно, нужно уменьшить эпсилон в 100 раз.
Поскольку ваши данные, по-видимому, являются координатами, вы должны использовать расстояние Хаверсин, потому что Земля не плоская, и выбирать эпсилон на основе расстояния, которое имеет смысл для вашей проблемы. Точное масштабирование может быть немного сложнее. Вероятно, это радианы, поэтому вам нужно преобразовать мили в радианы, чтобы перевести расстояния.