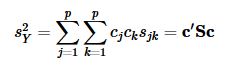Вычисление ковариации между 1-D массивами для включения в распространение неопределенности в Python
У меня есть четыре одномерных массива зависимых переменных. Они содержат сотни точек данных, но в этом примере я обрезал их до 20. Каждая точка представляет ячейку сетки на карте.
import numpy as np
A=np.asarray([0.195, 0.154, 0.208, 0.22, 0.204, 0.175, 0.184, 0.187, 0.171, 0.2, 0.222, 0.235, 0.206, 0.215, 0.222, 0.252, 0.269, 0.251, 0.285, 0.28])
B=np.asarray([0.119, 0.134, 0.132, 0.121, 0.11, 0.097, 0.13, 0.106, 0.103, 0.139, 0.124, 0.147, 0.152, 0.123, 0.177, 0.172, 0.18, 0.182, 0.197, 0.193])
C=np.asarray([0.11, 0.1, 0.103, 0.111, 0.105, 0.098, 0.099, 0.093, 0.105, 0.099, 0.113, 0.093, 0.104, 0.095, 0.099, 0.105, 0.108, 0.128, 0.125, 0.118])
D=np.asarray([-0.015, -0.015, -0.007, -0.02, 0.002, 0.009, 0.019, 0.0, -0.02, -0.001, -0.006, -0.015, -0.03, -0.036, -0.051, -0.058, -0.065, -0.081, -0.082, -0.055])
Ошибка каждой переменной содержится в массивах:
A_err=np.asarray([ 0.016, 0.015, 0.017, 0.016, 0.015, 0.016, 0.016, 0.018, 0.015, 0.014, 0.015, 0.016, 0.017, 0.016, 0.017, 0.017, 0.017, 0.017, 0.017, 0.017])
B_err=np.asarray([ 0.045, 0.049, 0.039, 0.044, 0.036, 0.027, 0.032, 0.033, 0.029, 0.036, 0.032, 0.027, 0.04 , 0.022, 0.034, 0.026, 0.021, 0.028, 0.035, 0.028])
C_err=np.zeros(20)+0.7
D_err=np.zeros(20)+0.9
Y является суммой A, B, C и D:
Y=A+B+C+D
Очевидно, что Y будет иметь различное значение в каждой из 20 ячеек сетки. То, что я пытаюсь вычислить, является ошибкой при каждом измерении Y. Первоначально я рассчитывал это просто как:
Y_err=np.sqrt(A_err**2 + B_err**2 + C_err**2 + D_err**2)
Но это не включает условия ковариации. Поскольку у меня нет выражений для переменных A, B, C и D в терминах друг друга, единственный способ увидеть ковариации - это использовать матрицу корреляции-ковариации:
X = np.vstack([A,B,C,D])
C = np.cov(X)
print(C)
[[ 1.31819737e-03 9.52921053e-04 2.17881579e-04 -8.58197368e-04]
[ 9.52921053e-04 9.87252632e-04 1.69478947e-04 -7.97089474e-04]
[ 2.17881579e-04 1.69478947e-04 9.47868421e-05 -1.88007895e-04]
[ -8.58197368e-04 -7.97089474e-04 -1.88007895e-04 8.85081579e-04]]
Мне неясно, могу ли я просто добавить каждый из шести недиагональных матричных членов:
σ_AB=9.52921053e-04
σ_AC=2.17881579e-04
σ_AD=-8.58197368e-04
σ_BC=1.69478947e-04
σ_BD=-7.97089474e-04
σ_CD=-1.88007895e-04
в уравнение для распространения ошибки, так что:
Y_err=np.sqrt(A_err**2 + B_err**2 + C_err**2 + D_err**2 + 2*σ_AB + 2*σ_AC + 2*σ_AD + 2*σ_BC + 2*σ_BD + 2*σ_CD)
Или это совершенно неверно?
1 ответ
Это то, что я понимаю о ваших измерениях: если у вас было несколько измерений для одной сетки. Тогда вы бы вычислили стандартную ошибку в измерениях из этих наблюдений, включая перекрестные слагаемые. Здесь у вас есть одно измерение для каждой сетки и ошибки, связанной с этим измерением. Поскольку вы измерили для всех 20 сеток одну и ту же процедуру, вы можете рассчитать std_err для вашей процедуры измерения и добавить одинаковую неопределенность для всех ваших 20 наблюдений.
Для моделирования неопределенностей в измерении ошибок (только в терминах ошибок), т.е. стандартной ошибки в Y_err. Ковариационные / перекрестные термины дают вам представление о том, как неопределенности / ошибки в измерениях связаны друг с другом (т.е. σ_A_err_B_err), а не как фактические переменные измерения связаны друг с другом (то есть σ_AB).
Вместо: X = np.vstack([A,B,C,D])
Тебе нужно: X = np.vstack([A_err,B_err,C_err,D_err]), чтобы рассчитать ковариацию в терминах ошибок.
Ваша формула должна быть: Все они содержатся в вашей ковариационной матрице (включая диагональные члены): np.cov(np.vstack([A_err,B_err,C_err,D_err]))
Y_std_err= np.sqrt(σ_A_err**2 + σ_B_err**2 + σ_C_err**2 + σ_D_err**2 +
2*σ_A_err_B_error + 2*σ_A_err_C_err + 2*σ_A_err_D_err +
2*σ_B_err_C_err + 2*σ_B_err_D_err + 2*σ_C_err_D_err)
Кроме того, вы можете напрямую умножить матрицы, чтобы получить выборочную дисперсию для y вместо формулы суммирования.
Где S, здесь - выборочная матрица дисперсии / ковариации для слагаемых ошибок, а c - коэффициенты линейной комбинации модели измерения.
X = np.vstack([A_err,B_err,C_err,D_err]) # matrix of error terms
cov = np.cov(X)
c = np.asarray([1, 1, 1, 1]) # coefficients: linear combination
var_y_err = np.matmul(c.T, np.matmul(cov,c))
np.sqrt(var_y_err) # standard error in measurement of Y: stdy_y_err
# 0.007379024325749306
Значения Y должны быть просто суммой A,B,C,D и каждая точка массива имеет выше (+ или -) среднее значение Y_error (в зависимости от направления ошибки), имея свою собственную стандартную ошибку (+/-).
Y = A + B + C + D (+ or -) Y_err_avg # Y_err will be an interval for every grid:
Y_err_avg = sum(A_err + B_err + C_err + D_err)/ 20 + (+/-) std_y_err
PS: я не эксперт по распространению ошибок. Я бы порекомендовал вам опубликовать свой запрос на перекрестной проверке, чтобы получить экспертное мнение.