Рассеянный график с лессовой линией, лессовая линия не должна отображаться в данном регионе
Я нанесу здесь значения по длине для хромосомы
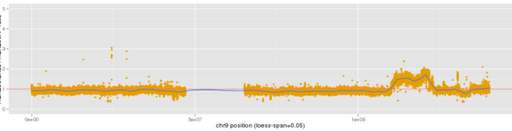
Средняя область без точек не содержит данных и не должна получать линию лесса. Как я могу изменить свой код, чтобы остановить линию лесса над этим регионом? Данные непрерывны, но я мог бы добавить строки, чтобы пометить пустую область каким-либо специальным значением или добавить столбец с меткой?? но как использовать это в команде?
моя текущая команда:
library(IDPmisc)
# plot settings (edit here)
spanv<-0.05
pointcol1="#E69F00"
pointcol2="#56B4E9"
pointcol3="#009E73"
points=20
linecol="green"
xlabs=paste(onechr, " position", " (loess-span=", spanv, ")", sep="")
data1<-NaRV.omit(data[,c(2,7)]) # keep only x and y for the relevant data
# and clean NA and Inf
ylabs='E / A - ratio'
p1<-ggplot(data1, aes(x=start, y=E.R)) +
ylim(0,5) +
geom_point(shape=points, col=pointcol1, na.rm=T) +
geom_hline(aes(yintercept=1, col=linecol)) +
geom_smooth(method="loess", span=spanv, fullrange=F, se=T, na.rm=T) +
xlab(xlabs) +
ylab(ylabs)
1 ответ
Решение
Я бы сделал одну из двух вещей:
- Сделать
loess()примерка снаружиggplot(), прогнозировать для двух регионов отдельно и добавлять каждый набор прогнозов к графику со своими собственнымиgeom_line()слой. - Как и выше, но на этот раз
ggplot()сфера деятельности. Добавьте два слоя на график, а не один, используя обаgeom_smooth(), но главное изменитьdataАргумент предоставляется каждому для ссылки только на одну или другую часть данных.
Для последнего, возможно, что-то вроде:
....
geom_smooth(data = data[1:n, ], method="loess", span=spanv, fullrange=FALSE,
se=TRUE, na.rm=TRUE) +
geom_smooth(data = data[m:k, ], method="loess", span=spanv, fullrange=FALSE,
se=TRUE, na.rm=TRUE)
....
где n а также m а также k обратитесь к индексам, которые отмечают конец набора 1 и начало и конец набора 2 и которые должны быть определены или предоставлены вами напрямую.