Как добавить внешнюю легенду в ggpairs()?
Я строю матрицу рассеяния, используя ggpairs, Я использую следующий код:
# Load required packages
require(GGally)
# Load datasets
data(state)
df <- data.frame(state.x77,
State = state.name,
Abbrev = state.abb,
Region = state.region,
Division = state.division
)
# Create scatterplot matrix
p <- ggpairs(df,
# Columns to include in the matrix
columns = c(3,5,6,7),
# What to include above diagonal
# list(continuous = "points") to mirror
# "blank" to turn off
upper = "blank",
legends=T,
# What to include below diagonal
lower = list(continuous = "points"),
# What to include in the diagonal
diag = list(continuous = "density"),
# How to label inner plots
# internal, none, show
axisLabels = "none",
# Other aes() parameters
colour = "Region",
title = "State Scatterplot Matrix"
)
# Show the plot
print(p)
и я получаю следующий сюжет:
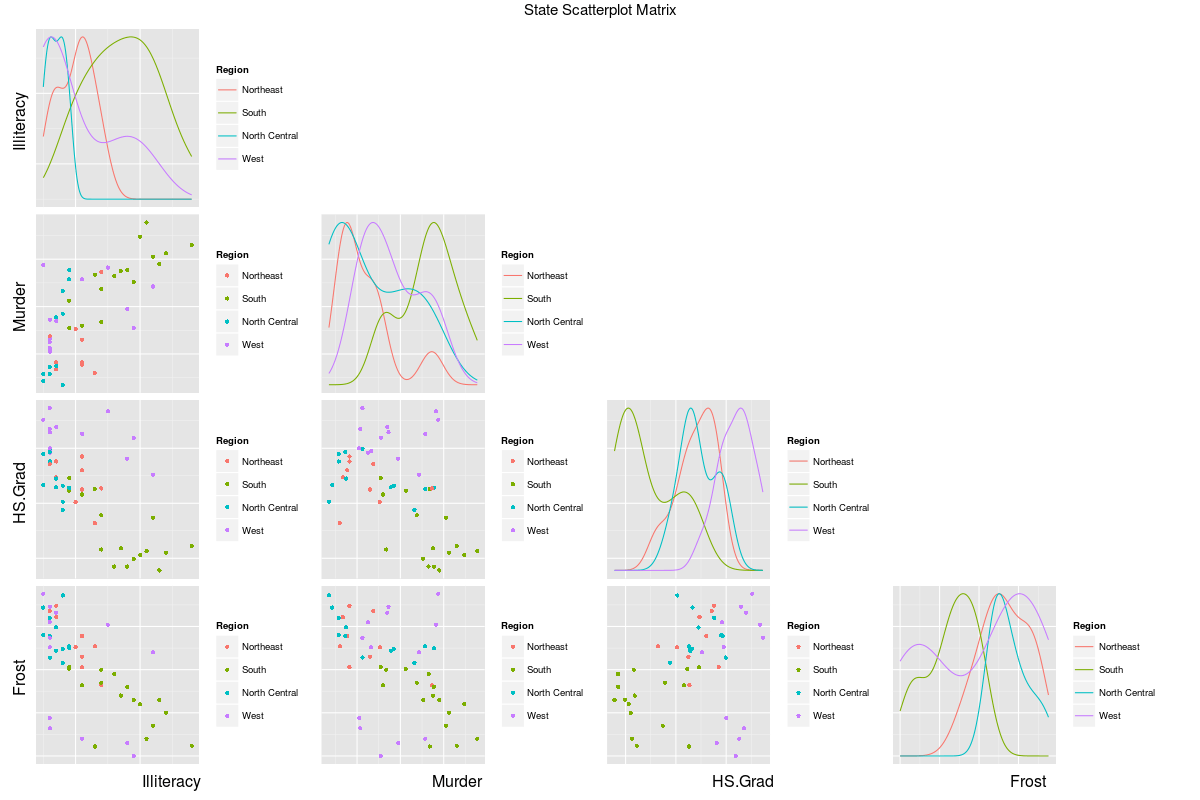
Теперь легко увидеть, что я получаю легенды для каждого графика в матрице. Я хотел бы иметь только одну универсальную легенду для всего сюжета. Как я могу это сделать? Любая помощь приветствуется.
2 ответа
Я работаю над чем-то похожим, я бы выбрал такой подход,
- Убедитесь, что легенды установлены на "ИСТИНА" в
ggpairsвызов функции Теперь итерируйте по вспомогательным участкам в матрице графиков и удалите легенды для каждого из них и просто оставьте один из них, так как все плотности построены в одном столбце.
colIdx <- c(3,5,6,7) for (i in 1:length(colIdx)) { # Address only the diagonal elements # Get plot out of matrix inner <- getPlot(p, i, i); # Add any ggplot2 settings you want (blank grid here) inner <- inner + theme(panel.grid = element_blank()) + theme(axis.text.x = element_blank()) # Put it back into the matrix p <- putPlot(p, inner, i, i) for (j in 1:length(colIdx)){ if((i==1 & j==1)){ # Move legend right inner <- getPlot(p, i, j) inner <- inner + theme(legend.position=c(length(colIdx)-0.25,0.50)) p <- putPlot(p, inner, i, j) } else{ # Delete legend inner <- getPlot(p, i, j) inner <- inner + theme(legend.position="none") p <- putPlot(p, inner, i, j) } } }
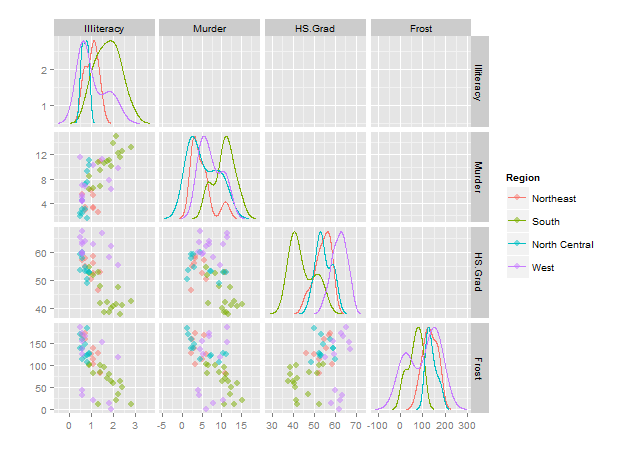
Надеюсь, кто-то покажет, как это можно сделать с ggpairs(...), Я бы хотел увидеть это сам. До тех пор, вот решение, которое не использует ggpairs(...)а ванильный ggplot с гранями.
library(ggplot2)
library(reshape2) # for melt(...)
library(plyr) # for .(...)
library(data.table)
xx <- with(df, data.table(id=1:nrow(df), group=Region, df[,c(3,5,6,7)]))
yy <- melt(xx,id=1:2, variable.name="H", value.name="xval")
setkey(yy,id,group)
ww <- yy[,list(V=H,yval=xval),key="id,group"]
zz <- yy[ww,allow.cartesian=T]
setkey(zz,H,V,group)
zz <- zz[,list(id, group, xval, yval, min.x=min(xval),min.y=min(yval),
range.x=diff(range(xval)),range.y=diff(range(yval))),by="H,V"]
d <- zz[H==V,list(x=density(xval)$x,
y=min.y+range.y*density(xval)$y/max(density(xval)$y)),
by="H,V,group"]
ggplot(zz)+
geom_point(subset= .(xtfrm(H)<xtfrm(V)),
aes(x=xval, y=yval, color=factor(group)),
size=3, alpha=0.5)+
geom_line(subset= .(H==V), data=d, aes(x=x, y=y, color=factor(group)))+
facet_grid(V~H, scales="free")+
scale_color_discrete(name="Region")+
labs(x="", y="")
Основная идея заключается в melt(...) ваш df в правильный формат для ggplot (xx), сделайте две копии (yy а также ww) и запустить декартово объединение на основе id а также group (Вот, id это просто номер строки и group это Region переменная), чтобы создать zz, Нам нужно рассчитать и масштабировать плотности извне (в таблице данных d). Несмотря на все это, он все еще работает быстрее, чем ggpairs(...),