Вращение 2-мерного массива с использованием NumPy без эффектов наложения
Я хотел бы повернуть только положительные значения пикселей в моем 2d-массиве на несколько градусов относительно центральной точки. Данные представляют концентрации аэрозоля из модели дисперсии шлейфа, а положение дымохода является источником вращения.
Я хотел бы повернуть этот образец рассеивания, учитывая направление ветра.
Концентрации сначала рассчитываются для направления ветра вдоль оси x, а затем переводятся в их повернутое положение, используя двухмерное линейное вращение вокруг центральной точки моего массива (положение дымохода) для всех точек, концентрация которых> 0. Вход X,Y к формуле вращения - это пиксельные индексы.
Моя проблема в том, что выходные данные являются псевдонимами, так как целые числа становятся числами с плавающей точкой. Чтобы получить целые числа, я округлял в большую или меньшую сторону результат. Однако это создает нулевые ячейки, которые становятся все более многочисленными с увеличением угла.
Может ли кто-нибудь помочь мне найти решение моей проблемы? Я хотел бы решить эту проблему, если это возможно, используя numpy или минимум пакетов...
Часть моего сценария, которая касается вычисления концентраций и поворота пикселя на 50° с.ш., следующая. Спасибо за помощь.
def linear2D_rotation(xcoord,ycoord,azimuth_degrees):
radians = (90 - azimuth_degrees) * (np.pi / 180) # in radians
xcoord_rotated = (xcoord * np.cos(radians)) - (ycoord * np.sin(radians))
ycoord_rotated = (xcoord * np.sin(radians)) + (ycoord * np.cos(radians))
return xcoord_rotated,ycoord_rotated
u_orient = 50 # wind orientation in degres from North
kernel = np.zeros((NpixelY, NpixelX)) # initialize matrix
Yc = int((NpixelY - 1) / 2) # position of central pixel
Xc = int((NpixelX - 1) / 2) # position of central pixel
nk = 0
for Y in list(range(0,NpixelX)):
for X in list(range(0,NpixelY)):
# compute concentrations only in positive x-direction
if (X-Xc)>0:
# nnumber of pixels to origin point (chimney)
dx = ((X-Xc)+1)
dy = ((Y-Yc)+1)
# distance of point to origin (chimney)
DX = dx*pixel_size_X
DY = dy*pixel_size_Y
# compute diffusivity coefficients
Sy, Sz = calcul_diffusivity_coeff(DX, stability_class)
# concentration at ground level below the centerline of the plume
C = (Q / (2 * np.pi * u * Sy * Sz)) * \
np.exp(-(DY / (2 * Sy)) ** 2) * \
(np.exp(-((Z - H) / (2 * Sz)) ** 2) + np.exp(-((Z + H) / (2 * Sz)) ** 2)) # at point away from center line
C = C * 1e9 # convert MBq to Bq
# rotate only if concentration value at pixel is positive
if C > 1e-12:
X_rot, Y_rot = linear2D_rotation(xcoord=dx, ycoord=dy,azimuth_degrees=u_orient)
X2 = int(round(Xc+X_rot))
Y2 = int(round(Yc-Y_rot)) # Y increases downwards
# pixels that fall out of bounds -> ignore
if (X2 > (NpixelX - 1)) or (X2 < 0) or (Y2 > (NpixelY - 1)):
continue
else:
# replace new pixel position in kernel array
kernel[Y2, X2] = C
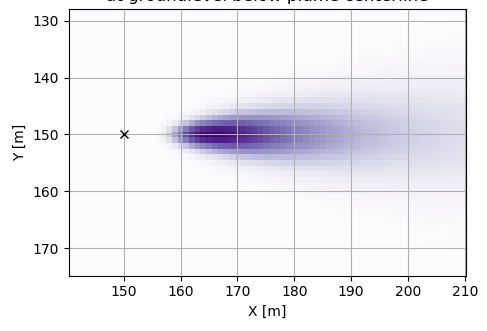
Исходный массив для поворота
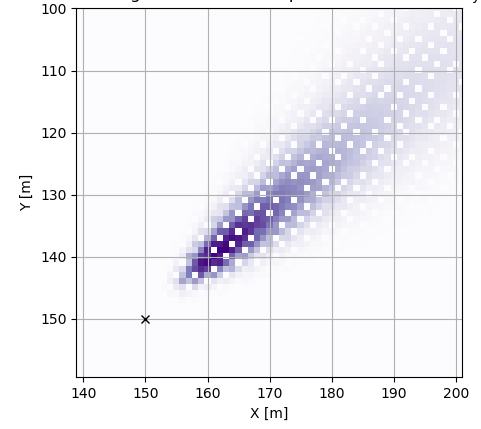
Вращенный массив на 40° с.ш., показывающий потерю данных
1 ответ
Ваше описание проблемы не совсем понятно, но вот несколько рекомендаций:
1.) Не изобретай велосипед. Существуют стандартные решения для таких вещей, как вращение пикселей. Используй их! В этом случае
scipy.ndimage.affine_transformдля выполнения вращения- однородная координатная матрица для задания поворота
- интерполяция ближайшего соседа (параметр
order=0в коде ниже).
2.) Не зацикливайтесь там, где это не нужно. Скорость, которую вы приобретаете, не обрабатывая неположительные пиксели, ничем не отличается от скорости, которую вы теряете в цикле. Скомпилированные функции могут перебирать множество избыточных нулей, прежде чем рукописный код Python догоняет их.
3.) Не ожидайте решения, которое отображает пиксели один в один, потому что это факт, что будут точки, которые не являются ближайшими соседями, и точки, которые являются ближайшими соседями, к множеству других точек. Имея это в виду, вы можете рассмотреть более гладкую интерполяцию более высокого порядка.
Сравнивая ваше решение со стандартным решением для инструментов, мы обнаруживаем, что последнее
дает сопоставимый результат гораздо быстрее и без этих дырочных артефактов.
Код (без прорисовки). Обратите внимание, что мне пришлось перенести и flipud чтобы выровнять результаты:
import numpy as np
from scipy import ndimage as sim
from scipy import stats
def mock_data(n, Theta=50, put_neg=True):
y, x = np.ogrid[-20:20:1j*n, -9:3:1j*n, ]
raster = stats.norm.pdf(y)*stats.norm.pdf(x)
if put_neg:
y, x = np.ogrid[-5:5:1j*n, -3:9:1j*n, ]
raster -= stats.norm.pdf(y)*stats.norm.pdf(x)
raster -= (stats.norm.pdf(y)*stats.norm.pdf(x)).T
return {'C': raster * 1e-9, 'Theta': Theta}
def rotmat(Theta, offset=None):
theta = np.radians(Theta)
c, s = np.cos(theta), np.sin(theta)
if offset is None:
return np.array([[c, -s] [s, c]])
R = np.array([[c, -s, 0], [s, c,0], [0,0,1]])
to, fro = np.identity(3), np.identity(3)
offset = np.asanyarray(offset)
to[:2, 2] = offset
fro[:2, 2] = -offset
return to @ R @ fro
def f_pp(C, Theta):
m, n = C.shape
clipped = np.maximum(0, 1e9 * data['C'])
clipped[:, :n//2] = 0
M = rotmat(Theta, ((m-1)/2, (n-1)/2))
return sim.affine_transform(clipped, M, order = 0)
def linear2D_rotation(xcoord,ycoord,azimuth_degrees):
radians = (90 - azimuth_degrees) * (np.pi / 180) # in radians
xcoord_rotated = (xcoord * np.cos(radians)) - (ycoord * np.sin(radians))
ycoord_rotated = (xcoord * np.sin(radians)) + (ycoord * np.cos(radians))
return xcoord_rotated,ycoord_rotated
def f_OP(C, Theta):
kernel = np.zeros_like(C)
m, n = C.shape
for Y in range(m):
for X in range(n):
if X > n//2:
c = C[Y, X] * 1e9
if c > 1e-12:
dx = X - n//2 + 1
dy = Y - m//2 + 1
X_rot, Y_rot = linear2D_rotation(xcoord=dx, ycoord=dy,azimuth_degrees=Theta)
X2 = int(round(n//2+X_rot))
Y2 = int(round(m//2-Y_rot)) # Y increases downwards
# pixels that fall out of bounds -> ignore
if (X2 > (n - 1)) or (X2 < 0) or (Y2 > (m - 1)):
continue
else:
# replace new pixel position in kernel array
kernel[Y2, X2] = c
return kernel
n = 100
data = mock_data(n, 70)
