Получение наблюдений в узле rpart (например, CART)
Я хотел бы проверить все наблюдения, которые достигли некоторого узла в дереве решений rpart. Например, в следующем коде:
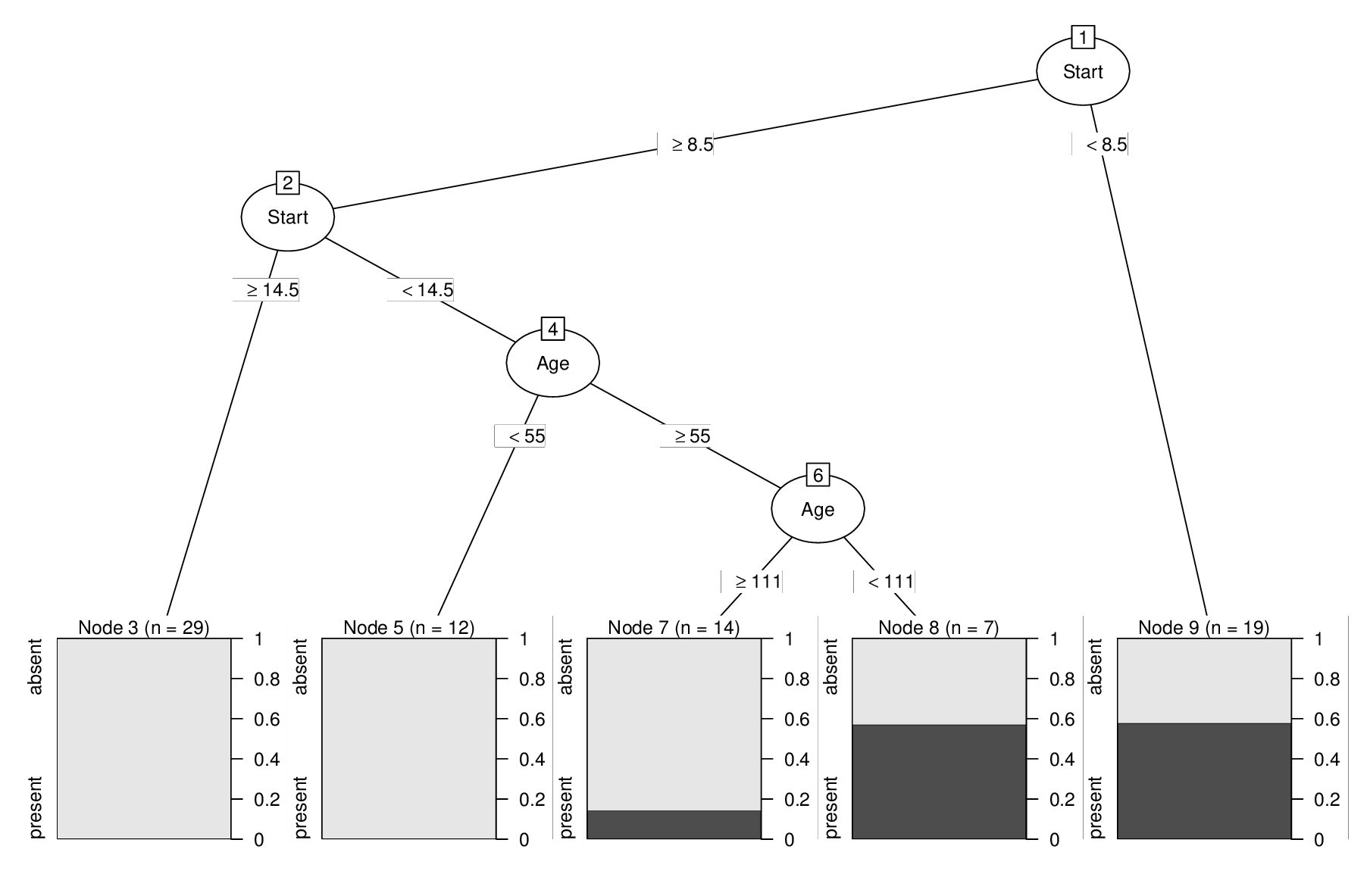
fit <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
fit
n= 81
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 81 17 absent (0.79012346 0.20987654)
2) Start>=8.5 62 6 absent (0.90322581 0.09677419)
4) Start>=14.5 29 0 absent (1.00000000 0.00000000) *
5) Start< 14.5 33 6 absent (0.81818182 0.18181818)
10) Age< 55 12 0 absent (1.00000000 0.00000000) *
11) Age>=55 21 6 absent (0.71428571 0.28571429)
22) Age>=111 14 2 absent (0.85714286 0.14285714) *
23) Age< 111 7 3 present (0.42857143 0.57142857) *
3) Start< 8.5 19 8 present (0.42105263 0.57894737) *
Я хотел бы видеть все наблюдения в узле (5) (то есть: 33 наблюдения, для которых Start>=8.5 и Start< 14.5). Очевидно, я мог бы вручную добраться до них. Но я хотел бы иметь некоторые функции, такие как (скажем) "get_node_date". Для которого я мог бы просто запустить get_node_date(5) - и получить соответствующие наблюдения.
Любые предложения о том, как это сделать?
6 ответов
Кажется, нет такой функции, которая позволяла бы извлекать наблюдения из определенного узла. Я бы решил это следующим образом: сначала определите, какие правила / ы используются для узла, в котором вы заинтересованы. Вы можете использовать path.rpart для этого. Затем вы можете применить правило / правила одно за другим, чтобы извлечь наблюдения.
Этот подход как функция:
get_node_date <- function(tree = fit, node = 5){
rule <- path.rpart(tree, node)
rule_2 <- sapply(rule[[1]][-1], function(x) strsplit(x, '(?<=[><=])(?=[^><=])|(?<=[^><=])(?=[><=])', perl = TRUE))
ind <- apply(do.call(cbind, lapply(rule_2, function(x) eval(call(x[2], kyphosis[,x[1]], as.numeric(x[3]))))), 1, all)
kyphosis[ind,]
}
Для узла 5 вы получаете:
get_node_date()
node number: 5
root
Start>=8.5
Start< 14.5
Kyphosis Age Number Start
2 absent 158 3 14
10 present 59 6 12
11 present 82 5 14
14 absent 1 4 12
18 absent 175 5 13
20 absent 27 4 9
23 present 96 3 12
26 absent 9 5 13
28 absent 100 3 14
32 absent 125 2 11
33 absent 130 5 13
35 absent 140 5 11
37 absent 1 3 9
39 absent 20 6 9
40 present 91 5 12
42 absent 35 3 13
46 present 139 3 10
48 absent 131 5 13
50 absent 177 2 14
51 absent 68 5 10
57 absent 2 3 13
59 absent 51 7 9
60 absent 102 3 13
66 absent 17 4 10
68 absent 159 4 13
69 absent 18 4 11
71 absent 158 5 14
72 absent 127 4 12
74 absent 206 4 10
77 present 157 3 13
78 absent 26 7 13
79 absent 120 2 13
81 absent 36 4 13
Через два года после первоначального поста, но может быть полезным для других. Назначения узлов для обучающих наблюдений в rpart можно получить из $where:
fit <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
fit$where
Как функция:
get_node <- function(rpart.object=fit, data=kyphosis, node.number=5) {
data[which(fit$where == node.number),]
}
get_node()
Это работает только для обучения наблюдений, но не для новых наблюдений.
rpart возвращает элемент rpart.object, который содержит необходимую информацию:
require(rpart)
fit2 <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
fit2
get_node_date <-function(nodeId,fit)
{
fit$frame[toString(nodeId),"n"]
}
for (i in c(1,2,4,5,10,11,22,23,3) )
cat(get_node_date(i,fit2),"\n")
Еще один способ - это найти все терминальные узлы любого конкретного узла и вернуть подмножество данных, использованных в вызове.
fit <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
head(subset.rpart(fit, 5))
# Kyphosis Age Number Start
# 2 absent 158 3 14
# 10 present 59 6 12
# 11 present 82 5 14
# 14 absent 1 4 12
# 18 absent 175 5 13
# 20 absent 27 4 9
subset.rpart <- function(tree, node = 1L) {
data <- eval(tree$call$data, parent.frame(1L))
wh <- sapply(as.integer(rownames(tree$frame)), parent)
wh <- unique(unlist(wh[sapply(wh, function(x) node %in% x)]))
data[rownames(tree$frame)[tree$where] %in% wh[wh >= node], ]
}
parent <- function(x) {
if (x[1] != 1)
c(Recall(if (x %% 2 == 0L) x / 2 else (x - 1) / 2), x) else x
}
partykit Пакет также предоставляет консервированное решение для этого. Вам просто нужно конвертировать rpart возражать против party класс, чтобы использовать его унифицированный интерфейс для работы с деревьями. И тогда вы можете использовать data_party() функция.
С использованием fit от вопроса и загрузив library("partykit") вы можете сначала принуждать rpart дерево к party:
pfit <- as.party(fit)
plot(pfit)
Есть только две маленькие неприятности для извлечения данных так, как вы хотите: (1) model.frame() Исходная посадка всегда сбрасывается при принуждении и требует повторного присоединения вручную. (2) Для узлов используется другая схема нумерации. Вы хотите узел 4 (а не 5) сейчас.
pfit$data <- model.frame(fit)
data4 <- data_party(pfit, 4)
dim(data4)
## [1] 33 5
head(data4)
## Kyphosis Age Start (fitted) (response)
## 2 absent 158 14 7 absent
## 10 present 59 12 8 present
## 11 present 82 14 8 present
## 14 absent 1 12 5 absent
## 18 absent 175 13 7 absent
## 20 absent 27 9 5 absent
Другой путь состоит в том, чтобы поднастроить поддерево, начиная с узла 4, а затем взять данные из этого
pfit4 <- pfit[4]
plot(pfit4)
затем data_party(pfit4) дает вам так же, как data4 выше. А также pfit4$data дает вам данные без (fitted) узел и предсказанный (response),
Альтернативный метод заключается в поиске всех дочерних узлов данного узла. Мы можем использоватьrpartобъект, чтобы найти их. Объединение этой информации с конечным узлом для каждой точки набора данных (кифоза в этом вопросе), полученного изfit$whereкак объяснил @rawar, вы можете получить все точки набора данных, участвующие в данном узле, не обязательно конечном.
Краткое изложение шагов:
- Найдите номера узлов и определите те, которые являются конечными узлами («листами»). Эту информацию можно найти в
frameэлемент объекта rpart. - Вычислить все дочерние узлы данного узла. Их можно вычислить рекурсивно, используя тот факт, что дочерними элементами узла являются
2*nи2*n+1, как поясняется вrpart.plotвиньетка об упаковке, стр. 26 - Как только листья свисают с узла
nизвестны, выберите точки в наборе данных на этих листьях, используяwhereэлемент объекта rpart
Я закодировал шаги 1 и 2 в функцииget_children_nodes()и шаг 3 в функцииget_node_data()это ответ на поставленный вопрос. В эту функцию я включил возможность распечатать соответствующее правило узла (rule = TRUE), чтобы получить тот же ответ, что и @datamineR
library(rpart)
library(rpart.plot)
fit <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
get_children_nodes <- function(tree, node){
# check if node is a leaf based in rpart object (tree) information (step 1)
z <- tree$frame
is_leaf <- z$var == "<leaf>"
nodes <- as.integer(row.names(z))
# find recursively all children nodes (step 2)
find_children <- function(node, nodes, is_leaf){
condition <- is_leaf[nodes == node]
if (condition) {
# If node is leaf, return it
v1 <- node
} else {
# If node is not leaf, search children leaf recursively
v1 <- c(find_children(2 * node, nodes, is_leaf),
find_children(2 * node + 1, nodes, is_leaf))
}
return(v1)
}
return(find_children(node, nodes, is_leaf))
}
get_node_data <- function(dataset, tree, node, rule = FALSE) {
# Find children nodes of the node
children_nodes <- get_children_nodes(tree, node)
# match those nodes into the rpart node identification
id_nodes <- which(as.integer(row.names(tree$frame)) %in% children_nodes)
# Get the elements in the datset involved in the node (step 3)
filtered_dataset <- dataset[tree$where %in% id_nodes, ]
# print the node rule if needed
if(rule) {
rpart::path.rpart(tree, node, pretty = TRUE)
cat(" \n")
}
return( filtered_dataset)
}
# Get the children nodes
get_children_nodes(fit, 5)
#> [1] 10 22 23
# Complete function to return the elements of node 5
get_node_data(kyphosis, fit, 5, rule = TRUE)
#>
#> node number: 5
#> root
#> Start>=8.5
#> Start< 14.5
#>
#> Kyphosis Age Number Start
#> 2 absent 158 3 14
#> 10 present 59 6 12
#> 11 present 82 5 14
#> 14 absent 1 4 12
#> 18 absent 175 5 13
#> 20 absent 27 4 9
#> 23 present 96 3 12
#> 26 absent 9 5 13
#> 28 absent 100 3 14
#> 32 absent 125 2 11
#> 33 absent 130 5 13
#> 35 absent 140 5 11
#> 37 absent 1 3 9
#> 39 absent 20 6 9
#> 40 present 91 5 12
#> 42 absent 35 3 13
#> 46 present 139 3 10
#> 48 absent 131 5 13
#> 50 absent 177 2 14
#> 51 absent 68 5 10
#> 57 absent 2 3 13
#> 59 absent 51 7 9
#> 60 absent 102 3 13
#> 66 absent 17 4 10
#> 68 absent 159 4 13
#> 69 absent 18 4 11
#> 71 absent 158 5 14
#> 72 absent 127 4 12
#> 74 absent 206 4 10
#> 77 present 157 3 13
#> 78 absent 26 7 13
#> 79 absent 120 2 13
#> 81 absent 36 4 13
Создано 14 августа 2023 г. с использованием reprex v2.0.2.