Моделирование влияния узлов на другие узлы
моделирование miRNA - генных взаимодействий
Мы заинтересованы в моделировании влияния изменения одного узла на другие узлы. Вероятно, нам нужно применить широту первого подхода (map_bfs в Tidygraph), чтобы рассчитать такие эффекты, но мы не можем понять это. Мы проверили алгоритмы потока или модели структурных уравнений ( SEM), но снова не смогли вписать их в нашу проблему.
Просто чтобы прояснить ситуацию, вот проблема в пошаговом объяснении. Пожалуйста, не позволяйте биологии напугать вас, пожалуйста, рассмотрите проблему в контексте сетевого анализа.
Шаг 1
MiRNA может связывать мРНК (кодируется геном) и истощать ее количество.
Шаг 2
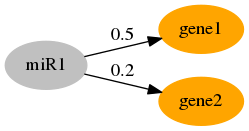
Одна miRNA контролирует много мРНК (только две, показанные на диаграмме, кодируются геном 1 и геном 2), из-за последовательностей семян и их типов эффективность истощения может варьироваться. Это может быть "сильный" или "слабый". На диаграмме ниже прочность показана как вес ребра. Эффективность истощения означает, сколько (по количеству или по соотношению) микроРНК связывается с этой конкретной мРНК. В небольшом образце, представленном ниже, 0,5 единицы доступного miR1 изолируются мРНК гена 1, а 0,2 единицы miR1 изолируются мРНК гена 2.
Шаг 3
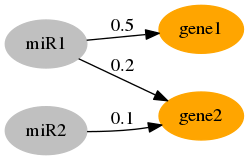
Теперь все усложняется, мРНК контролируется несколькими микроРНК, и они имеют разный вес из-за последовательностей семян.
Вопрос
Таким образом, как мы можем увидеть влияние изменения уровня мРНК гена 1 на уровень мРНК гена 2? Например, если мРНК гена 1 увеличилась в 2 раза, сколько единиц мРНК гена 2 увеличилось бы? Мы сказали "увеличение", потому что, когда мРНК гена 1 увеличивается, она будет секвестрировать больше miR1, следовательно, оставляя меньше доступного miR1 для связывания с мРНК гена 2.
Образец сети
example <- data.frame(mirna = c("miR1", "miR1", "miR2"),
gene = c("gene1","gene2", "gene2"),
seed= c("strong", "weak", "weak"),
seed_weight = c(0.5, 0.2, 0.1))
Много узлов
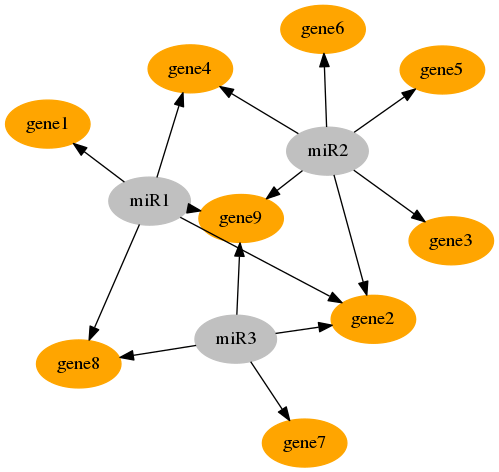
В приведенном выше примере мы представили меньшую сеть. Фактическая сеть была бы намного больше (сотни микроРНК и тысячи мРНК). Мы заинтересованы в расчете эффекта изменения одного узла в других узлах. Например, согласно приведенной ниже сети (весовые коэффициенты не показаны), если мРНК gene2 снижается в определенном размере, каково будет изменение уровней мРНК всех генов, учитывая тот факт, что изменения в секвестрации и доступности miR1, miR2 и miR3