Добавление информации в дерево - Rpart
Я хочу добавить информацию в мое дерево. Скажем, например, у меня есть такая база данных:
library(rpart)
library(rpart.plot)
set.seed(1)
mydb<-data.frame(results=rnorm(1000,0,1),expo=runif(1000),var1=sample(LETTERS[1:4],1000,replace=T),
var2=sample(LETTERS[5:6],1000,replace=T),var3=sample(LETTERS[20:25], 1000,replace=T))
Я могу запустить дерево:
mytree<-rpart(results~var1+var2+var3,data=mydb,cp=0)
pfit<- prune(mytree, cp=mytree$cptable[4,"CP"])
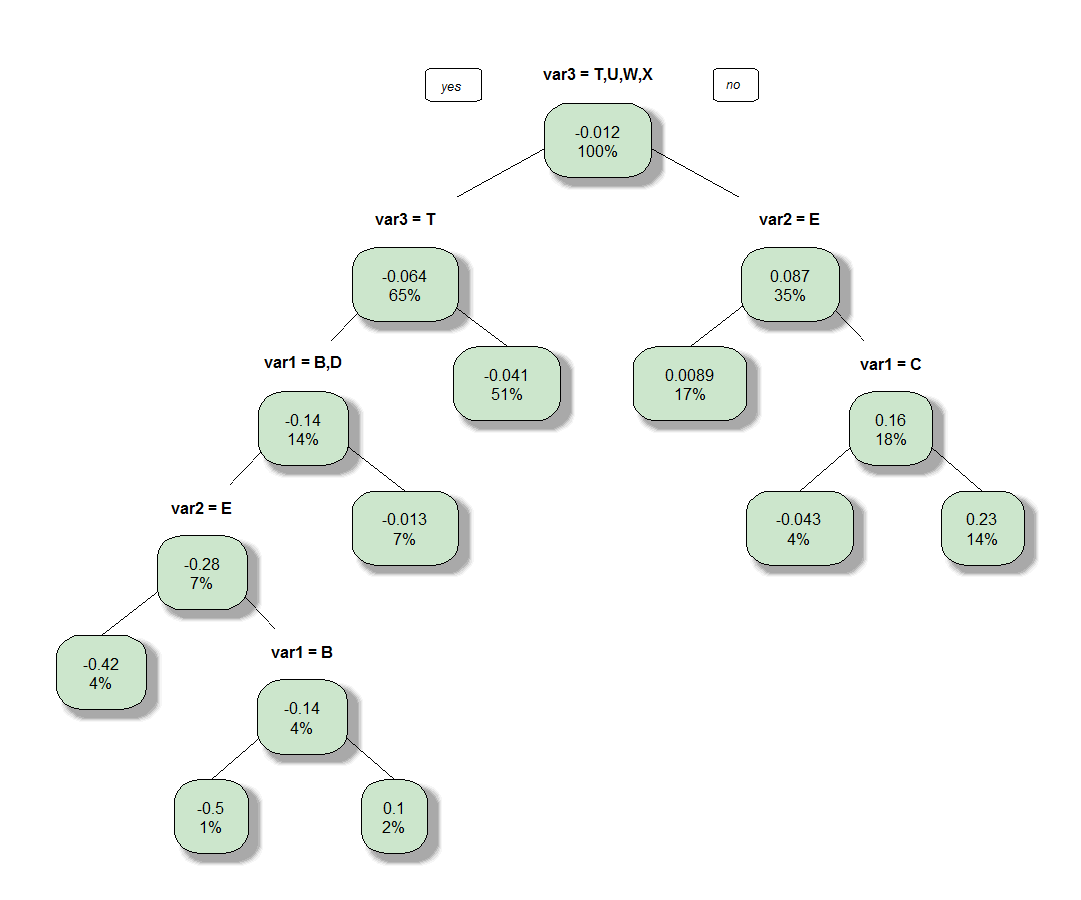
prp(pfit,type=1,extra=100,fallen.leaves=F,shadow.col="darkgray",box.col=rgb(0.8,0.9,0.8))
И это нормально для меня, но давайте представим, что я хочу знать среднюю экспозицию для каждого листа.
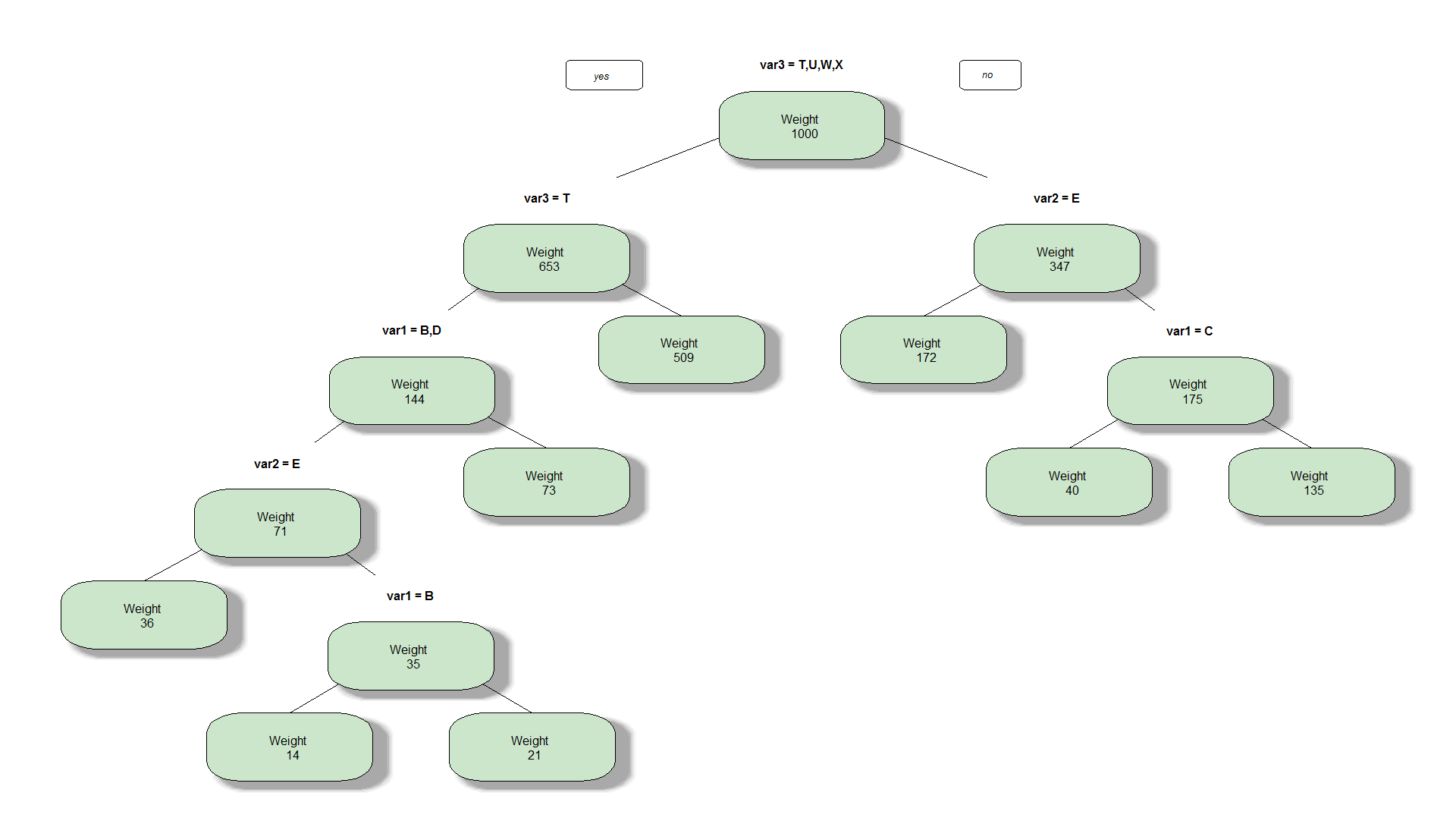
Я знаю, что могу добавить некоторую информацию в prp, например, вес каждого листа с функцией:
node.fun1 <- function(x, labs, digits, varlen)
{
paste("Weight \n",x$frame$wt)
}
prp(pfit,type=1,extra=100,fallen.leaves=F,shadow.col="darkgray",box.col=rgb(0.8,0.9,0.8),node.fun = node.fun1)
Но это работает, только если он рассчитан в кадре, результаты функции rpart.
Мой вопрос:
Как добавить пользовательскую информацию на график, например, среднюю экспозицию, или любую другую функцию, которая рассчитывает пользовательские индикаторы и добавляет ее в таблицу frame?
1 ответ
Это действительно хорошо, я не знал, что это был вариант.
Кажется, что вся работа заключается в получении подмножества исходных данных, используемых на каждом узле. Это легко для терминальных узлов, но я не нашел прямого способа идентифицировать строки данных, которые использовались в каждом узле, а не только в листьях. Если кто-то знает более простой способ, я хотел бы услышать это.
library('rpart.plot')
set.seed(1)
mydb<-data.frame(results=rnorm(1000,0,1),expo=runif(1000),var1=sample(LETTERS[1:4],1000,replace=T),
var2=sample(LETTERS[5:6],1000,replace=T),var3=sample(LETTERS[20:25], 1000,replace=T))
mytree<-rpart(results~var1+var2+var3,data=mydb,cp=0)
pfit<- prune(mytree, cp=mytree$cptable[4,"CP"])
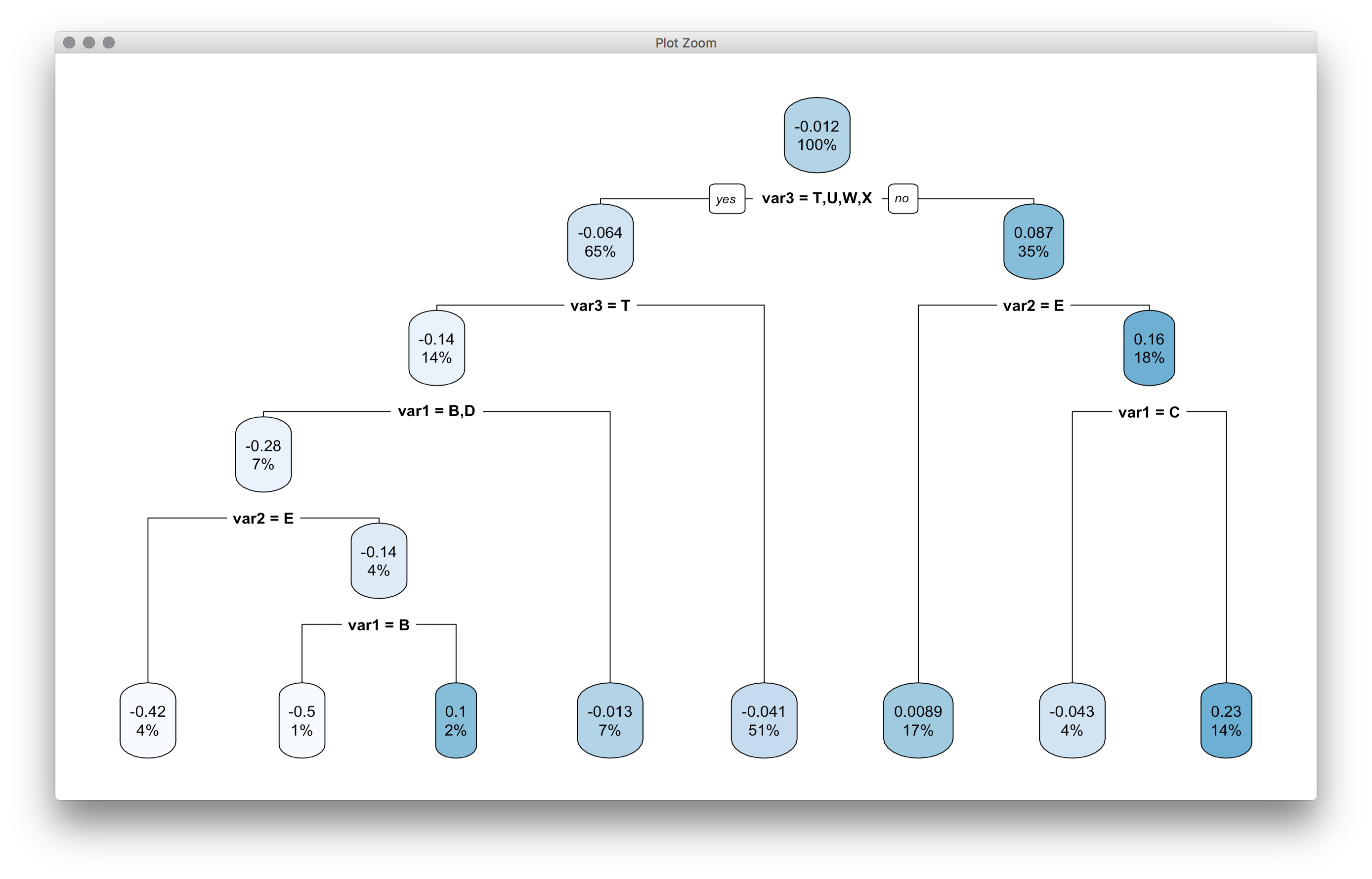
rpart.plot(pfit)
Определите вашу новую функцию, которая принимает x, результат примерки rpart (Я не смотрел на другие аргументы, но виньетка должна быть полезной).
Для каждой строки x$frame нам нужно получить данные, используемые для расчета сводной статистики. К несчастью, x$where только говорит нам конечный узел, в котором лежит каждое наблюдение. Поэтому для каждого номера узла мы используем subset.rpart чтобы получить базовые данные и делать с ними все, что вы хотите
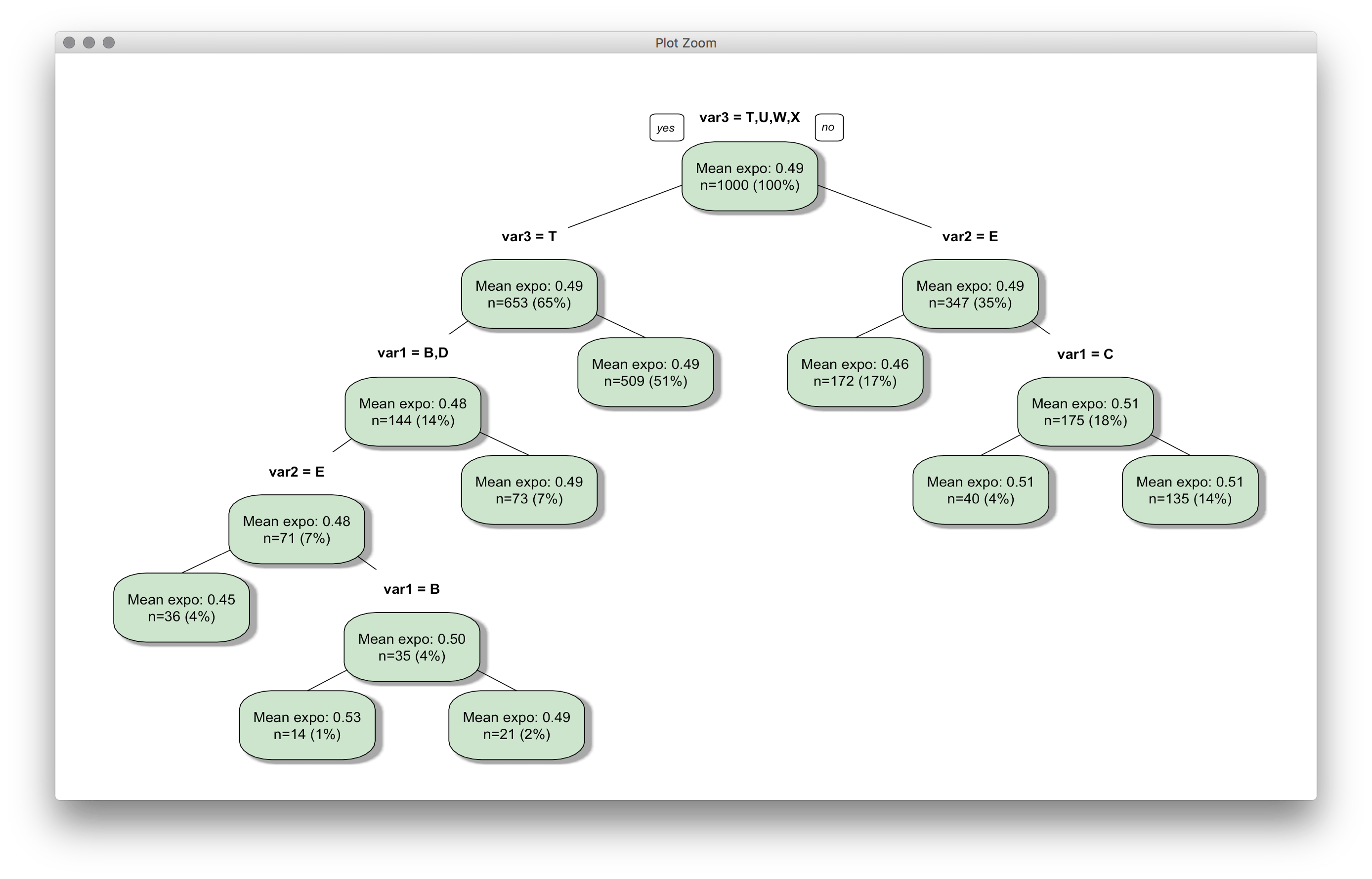
f <- function(x, labs, digits, varlen) {
nodes <- as.integer(rownames(x$frame))
z <- sapply(nodes, function(y) {
data <- subset.rpart(x, y)
c(mean = mean(data$expo), nrow(data), nrow(data) / length(x$where) * 100)
})
sprintf('Mean expo: %.2f\nn=%.0f (%.0f%%)', z[1, ], z[2, ], z[3, ])
}
prp(pfit, type=1, extra=100, fallen.leaves=FALSE,
shadow.col="darkgray", box.col=rgb(0.8,0.9,0.8),
node.fun = f)
Работа выполнена subset.rpart который принимает номер узла и возвращает подмножество data используется на узле.
subset.rpart <- function(tree, node = 1L) {
## returns subset of tree$call$data used on any node
data <- eval(tree$call$data, parent.frame(1L))
wh <- sapply(as.integer(rownames(tree$frame)), parent)
wh <- unique(unlist(wh[sapply(wh, function(x) node %in% x)]))
data[rownames(tree$frame)[tree$where] %in% wh[wh >= node], ]
}
parent <- function(x) {
## returns vector of parent nodes
if (x[1] != 1)
c(Recall(if (x %% 2 == 0L) x / 2 else (x - 1) / 2), x) else x
}
тесты
## tests
dim(subset.rpart(pfit, 1)) == dim(mydb)
# [1] TRUE TRUE
## terminal nodes
nodes <- as.integer(rownames(pfit$frame[pfit$frame$var %in% '<leaf>', ]))
sum(sapply(nodes, function(x) nrow(subset.rpart(pfit, x)))) == nrow(mydb)
# [1] TRUE
Я не знаю, если это именно то, что вы хотите, но попробуйте пакеты "sparkline" и "visNetwork". Они работают с объектами rpart